WikiDer > Методы анализа микрочипов
Методы анализа микрочипов используются при интерпретации данных, полученных в результате экспериментов с ДНК (Анализ генного чипа), РНК и белок микрочипы, которые позволяют исследователям исследовать состояние экспрессии большого количества генов - во многих случаях всего организма геном - в одном эксперименте.[нужна цитата] Такие эксперименты могут генерировать очень большие объемы данных, позволяя исследователям оценить общее состояние клетки или организма. Данные в таких больших объемах трудно - если вообще возможно - анализировать без помощи компьютерных программ.
Вступление
Анализ данных микроматрицы - это последний этап считывания и обработки данных, производимых микрочипом. Образцы проходят различные процессы, включая очистку и сканирование с помощью микрочипа, который затем производит большой объем данных, требующих обработки с помощью компьютерного программного обеспечения. Он включает в себя несколько отдельных шагов, как показано на изображении ниже. Изменение любого из шагов приведет к изменению результата анализа, поэтому проект MAQC[1] был создан для определения набора стандартных стратегий. Существуют компании, которые используют протоколы MAQC для выполнения полного анализа.[2]
Методы

Большинство производителей микрочипов, таких как Affymetrix и Agilent,[3] поставляют коммерческое программное обеспечение для анализа данных вместе со своими продуктами на основе микрочипов. Существуют также варианты с открытым исходным кодом, в которых используются различные методы анализа данных микрочипов.
Агрегация и нормализация
Сравнение двух разных наборов или двух разных образцов, гибридизованных с одним и тем же набором, обычно включает внесение поправок на систематические ошибки, вызванные различиями в процедурах и эффектами интенсивности красителя. Нормализация красителя для двух цветовых массивов часто достигается локальная регрессия. LIMMA предоставляет набор инструментов для коррекции фона и масштабирования, а также возможность усреднять повторяющиеся пятна на слайде.[4] Распространенный метод оценки того, насколько хорошо нормализован массив, - это построить Сюжет MA данных. Графики MA могут быть созданы с использованием таких программ и языков, как R, MATLAB и Excel.
Необработанные данные Affy содержат около двадцати зондов для одной и той же РНК-мишени. Половина из них - это «пятна несоответствия», которые не точно соответствуют целевой последовательности. Теоретически они могут измерить количество неспецифического связывания для данной мишени. Устойчивое среднее значение по нескольким массивам (RMA) [5] - это подход к нормализации, который не использует преимущества этих пятен несоответствия, но все же должен суммировать идеальные совпадения через средний полироль.[6] Алгоритм медианной полировки, хотя и надежен, ведет себя по-разному в зависимости от количества проанализированных образцов.[7] Квантильная нормализация, также являющаяся частью RMA, является одним из разумных подходов к нормализации пакета массивов, чтобы сделать дальнейшие сравнения значимыми.
Текущий алгоритм Affymetrix MAS5, в котором используются как датчики идеального совпадения, так и датчики несовпадения, по-прежнему пользуется популярностью и демонстрирует хорошие результаты в тестах лицом к лицу.[8]
Факторный анализ для надежного суммирования микрочипов (FARMS)[9] представляет собой основанный на модели метод суммирования данных массива на уровне датчика точного совпадения. Он основан на модели факторного анализа, для которой апостериорный метод байесовского максимума оптимизирует параметры модели в предположении гауссовского шума измерения. Согласно тесту Affycomp[10] FARMS превзошла все другие методы обобщения по чувствительности и специфичности.
Выявление значимого дифференциального выражения
Существует множество стратегий для идентификации массивов зондов, которые показывают необычный уровень сверхэкспрессии или недостаточной экспрессии. Самый простой - назвать «значимым» любой зонд, который отличается в среднем как минимум в два раза между группами лечения. Более сложные подходы часто связаны с t-тесты или другие механизмы, учитывающие как размер эффекта, так и его изменчивость. Любопытно, что p-значения, связанные с конкретными генами, плохо воспроизводятся между повторными экспериментами, и списки, созданные прямым кратным изменением, работают намного лучше.[11][12] Это чрезвычайно важное наблюдение, поскольку цель проведения экспериментов связана с предсказанием общего поведения. Группа MAQC рекомендует использовать оценку кратности изменения плюс нестрогую отсечку p-значения, дополнительно указывая на то, что изменения в фоновой коррекции и процессе масштабирования оказывают лишь минимальное влияние на порядок ранжирования различий в кратности изменения, но существенно влияют на p-значения.
Кластеризация
Кластеризация - это метод интеллектуального анализа данных, используемый для группировки генов с похожими паттернами экспрессии. Иерархическая кластеризация, и k-означает кластеризацию широко используемые методы в анализе микрочипов.
Иерархическая кластеризация
Иерархическая кластеризация - это статистический метод поиска относительно однородный кластеры. Иерархическая кластеризация состоит из двух отдельных фаз. Первоначально матрица расстояний содержащий все попарные расстояния между генами. Корреляция Пирсона и Корреляция Спирмена часто используются как оценки несходства, но другие методы, например Манхэттенское расстояние или же Евклидово расстояние, также может применяться. Учитывая количество доступных мер расстояния и их влияние на результаты алгоритма кластеризации, в нескольких исследованиях сравнивались и оценивались различные меры расстояния для кластеризации данных микрочипа с учетом их внутренних свойств и устойчивости к шуму.[13][14][15] После вычисления исходной матрицы расстояний алгоритм иерархической кластеризации либо (A) итеративно объединяет два ближайших кластера, начиная с отдельных точек данных (агломерационный, восходящий подход, который довольно часто используется), либо (B) итеративно разделяет кластеры. начиная с комплектации (спорный, нисходящий подход). После каждого шага новая матрица расстояний между вновь сформированными кластерами и другими кластерами пересчитывается. К методам иерархического кластерного анализа относятся:
- Одиночная связь (минимальный метод, ближайший сосед)
- Средняя связь (UPGMA).
- Полная связь (максимальный метод, самый дальний сосед)
Различные исследования уже эмпирически показали, что алгоритм кластеризации с одним сцеплением дает плохие результаты при использовании для данных микроматрицы экспрессии генов, и поэтому его следует избегать.[15][16]
К-средство кластеризации
Кластеризация K-средних - это алгоритм для группировки генов или образцов на основе шаблона в K группы. Группировка осуществляется путем минимизации суммы квадратов расстояний между данными и соответствующим кластером. центроид. Таким образом, цель кластеризации K-средних - классифицировать данные на основе аналогичного выражения.[17] Алгоритм кластеризации K-средних и некоторые его варианты (в том числе k-medoids) показали хорошие результаты для данных экспрессии генов (по крайней мере, лучше, чем методы иерархической кластеризации). Эмпирические сравнения k-означает, k-medoids, иерархические методы и различные меры расстояния можно найти в литературе.[15][16]
Распознавание образов
Коммерческие системы для анализа генных сетей, такие как Ingenuity[18] и студия Pathway[19] создавать визуальные представления дифференциально экспрессируемых генов на основе современной научной литературы. Некоммерческие инструменты, такие как FunRich,[20] GenMAPP и Moksiskaan также помогают в организации и визуализации данных генной сети, полученных в результате одного или нескольких экспериментов с микрочипами. Широкий спектр инструментов анализа микрочипов доступен через Биокондуктор написано в Язык программирования R. Часто цитируемый модуль SAM и другие инструменты микрочипов[21] доступны через Стэнфордский университет. Другой набор можно приобрести в Гарварде и Массачусетском технологическом институте.[22]
Также были разработаны специализированные программные инструменты для статистического анализа для определения степени чрезмерной или недостаточной экспрессии гена в эксперименте с микрочипом относительно эталонного состояния, чтобы помочь в идентификации генов или наборов генов, связанных с конкретными фенотипы. Один из таких методов анализа, известный как Обогащение генетического набора Анализ (GSEA), использует Колмогоров-Смирновстатистика стиля для определения групп генов, которые регулируются вместе.[23] Этот сторонний статистический пакет предлагает пользователю информацию об интересующих генах или наборах генов, включая ссылки на записи в базах данных, таких как NCBI. GenBank и кураторские базы данных, такие как Biocarta[24] и Генная онтология. Инструмент анализа обогащения белковых комплексов (COMPLEAT) обеспечивает аналогичный анализ обогащения на уровне белковых комплексов.[25] Инструмент может идентифицировать динамическую регуляцию белкового комплекса в различных условиях или временных точках. Связанная система, КРАСКА[26] и ОБЪЕМ[27] выполняет статистический анализ промоторных областей генов, выявляя избыточное и недостаточное представление ранее идентифицированных фактор транскрипции элементы ответа. Еще один инструмент статистического анализа - это статистика сумм рангов для коллекций наборов генов (RssGsc), которая использует функции распределения вероятностей суммы рангов для поиска наборов генов, объясняющих экспериментальные данные.[28] Еще один подход - контекстный метаанализ, то есть выяснение того, как кластер генов реагирует на различные экспериментальные контексты. Исследователь генов это общедоступный инструмент для выполнения контекстного метаанализа по таким контекстам, как анатомические части, стадии развития и реакция на болезни, химические вещества, стрессы и новообразования.
Анализ значимости микрочипов (SAM)
Анализ значимости микрочипов (SAM) это статистический метод, основанная в 2001 году Вирджинией Тушер, Роберт Тибширани и Гилберт Чу, для определения, есть ли изменения в экспрессия гена статистически значимы. С появлением ДНК-микрочипы, теперь можно измерить экспрессию тысяч генов в одном эксперименте по гибридизации. Сгенерировано много данных, и метод сортировки того, что важно, а что нет, очень важен. SAM распространяется Стэндфордский Университет в R-пакет.
SAM идентифицирует статистически значимые гены, выполняя специфичные для генов t-тесты и вычисляет статистику dj для каждого гена j, который измеряет силу связи между экспрессией гена и переменной ответа.[29][30][31] Этот анализ использует непараметрическая статистика, поскольку данные могут не соответствовать нормальное распределение. Переменная ответа описывает и группирует данные на основе экспериментальных условий. В этом методе повторяется перестановки данных используются для определения того, является ли экспрессия какого-либо гена значимой для ответа. Использование анализа на основе перестановок учитывает корреляции в генах и позволяет избежать параметрический предположения о распределении отдельных генов. Это преимущество перед другими методами (например, ANOVA и Бонферрони), которые предполагают равную дисперсию и / или независимость генов.[32]
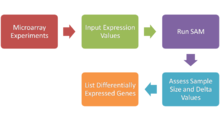
Базовый протокол
- Выполнять микрочип эксперименты - ДНК-микрочип с олиго- и кДНК-праймерами, массивы SNP, массивы белков и т. д.
- Анализ входных выражений в Microsoft Excel - см. Ниже
- Запуск SAM как надстройки Microsoft Excel
- Отрегулируйте параметр настройки дельты, чтобы получить значительное количество генов вместе с приемлемой частотой ложного обнаружения (FDR) и оценить размер выборки, вычислив среднюю разницу в экспрессии в контроллере графиков SAM
- Список дифференциально экспрессируемых генов (положительно и отрицательно экспрессируемых генов)
Запуск SAM
- SAM доступен для загрузки в Интернете по адресу http://www-stat.stanford.edu/~tibs/SAM/ для академических и неакадемических пользователей после завершения этапа регистрации.
- SAM запускается как надстройка Excel, а контроллер графика SAM позволяет настраивать частоту ложного обнаружения и дельту, в то время как функции вывода SAM и вывода SAM создают список важных генов, таблицу дельты и оценку размеров выборки.
- Перестановки рассчитываются исходя из количества образцов
- Блокировать перестановки
количество перестановок задается пользователем при вводе правильных значений для набора данных для запуска SAM
Форматы ответов
Типы:[29]
- Количественный - с реальной оценкой (например, пульс)
- Один класс - проверяет, отличается ли средняя экспрессия гена от нуля
- Два класса - два набора измерений
- Непарный - единицы измерения в двух группах разные; например контрольная и лечебная группы с образцами от разных пациентов
- Парный - одинаковые экспериментальные единицы измеряются в двух группах; например образцы до и после лечения от одних и тех же пациентов
- Мультикласс - более двух групп, каждая из которых содержит разные экспериментальные единицы; обобщение двухклассового непарного типа
- Выживание - данные о времени до события (например, смерть или рецидив)
- Курс времени - каждая экспериментальная единица измеряется более чем в одной временной точке; экспериментальные единицы делятся на одно- или двухклассные конструкции
- Открытие паттернов - не указан явный параметр ответа; пользователь указывает собственный ген (главный компонент) данных выражения и рассматривает его как количественный ответ
Алгоритм
SAM вычисляет тестовую статистику для относительной разницы в экспрессии генов на основе анализа перестановок данных экспрессии и вычисляет частоту ложного обнаружения. Основные расчеты программы проиллюстрированы ниже.[29][30][31]
В sо константа выбрана так, чтобы минимизировать коэффициент вариации dя. ря равно уровню экспрессии (x) гена я в условиях эксперимента.
Свернуть изменения (t) указаны для гарантии того, что гены вызывают значительное изменение, по крайней мере, на заранее заданное количество. Это означает, что абсолютное значение средних уровней экспрессии гена при каждом из двух условий должно быть больше кратного изменения (t), чтобы называться положительным, и меньше, чем обратное кратное изменение (t), чтобы называться отрицательным.
Алгоритм SAM можно сформулировать как:
- Заказать статистику испытаний по величине [30][31]
- Для каждой перестановки вычислить упорядоченные нулевые (незатронутые) оценки [30][31]
- Постройте график упорядоченной статистики теста против ожидаемых нулевых оценок [30][31]
- Считайте каждый ген значимым, если абсолютное значение тестовой статистики для этого гена за вычетом средней тестовой статистики для этого гена превышает установленный порог. [31]
- Оцените частоту ложного обнаружения на основе ожидаемых и наблюдаемых значений [30][31]
Выход
- Значимые наборы генов
- Положительный набор генов - более высокая экспрессия большинства генов в наборе генов коррелирует с более высокими значениями фенотипа у
- Отрицательный набор генов - более низкая экспрессия большинства генов в наборе генов коррелирует с более высокими значениями фенотипа у
Возможности SAM
- Данные из Oligo или массивов кДНК, массива SNP, массивов белков и т. Д. может использоваться в SAM[30][31]
- Сопоставляет данные экспрессии с клиническими параметрами[32]
- Коррелирует данные выражения со временем[29]
- Использует перестановку данных для оценки уровня ложного обнаружения для множественного тестирования[30][31][32][35]
- Сообщает о локальной частоте ложного обнаружения (FDR для генов, имеющих схожую dя как этот ген)[29] и пропустить ставки [29][30]
- Может работать с блокированным дизайном, когда обработки применяются в разных пакетах массивов[29]
- Может регулировать порог, определяющий количество гена, называемого значимым[29]
Исправление ошибок и контроль качества
Контроль качества
Целые массивы могут иметь очевидные недостатки, обнаруживаемые визуальным осмотром, попарными сравнениями с массивами в той же экспериментальной группе или анализом деградации РНК.[36] Результаты можно улучшить, полностью исключив эти массивы из анализа.
Коррекция фона
В зависимости от типа массива сигнал, относящийся к неспецифическому связыванию флуорофора, можно вычесть для достижения лучших результатов. Один из подходов заключается в вычитании средней интенсивности сигнала в области между пятнами. Различные инструменты для коррекции фона и дальнейшего анализа доступны от TIGR,[37] Agilent (GeneSpring),[38] и Решения Ocimum Bio (Геновиз).[39]
Точечная фильтрация
Визуальная идентификация местных артефактов, таких как дефекты печати или стирки, также может предполагать удаление отдельных пятен. Это может занять значительное время в зависимости от качества изготовления массива. Кроме того, некоторые процедуры требуют удаления всех пятен со значением экспрессии ниже определенного порога интенсивности.
Смотрите также
Рекомендации
- ^ Доктор Леминг Ши, Национальный центр токсикологических исследований. «Проект контроля качества MicroArray (MAQC)». Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США. Получено 2007-12-26.
- ^ «GenUs BioSystems - Услуги - Анализ данных». Получено 2008-01-02.
- ^ «Agilent | ДНК-микрочипы». Архивировано из оригинал 22 декабря 2007 г.. Получено 2008-01-02.
- ^ «Библиотека LIMMA: линейные модели для данных микрочипов». Получено 2008-01-01.
- ^ Иризарри, РА; Хоббс, В; Коллин, Ф; Бизер-Барклай, Ю. Д.; Антонеллис, KJ; Scherf, U; Скорость, ТП (2003). «Исследование, нормализация и обобщение данных уровня зондов с массивом олигонуклеотидов высокой плотности». Биостатистика. 4 (2): 249–64. Дои:10.1093 / биостатистика / 4.2.249. PMID 12925520.
- ^ Bolstad BM, Irizarry RA, Astrand M, Speed TP (2003). «Сравнение методов нормализации для данных массива олигонуклеотидов высокой плотности на основе дисперсии и смещения». Биоинформатика. 19 (2): 185–93. Дои:10.1093 / биоинформатика / 19.2.185. PMID 12538238.
- ^ Георгий FM, Болджер AM, Lohse M, Usadel B (2010). «Артефакты, управляемые алгоритмами, в срединном полированном обобщении данных Microarray». BMC Биоинформатика. 11: 553. Дои:10.1186/1471-2105-11-553. ЧВК 2998528. PMID 21070630.
- ^ Лим В.К., Ван К., Лефевр С., Калифано А (2007). «Сравнительный анализ процедур нормализации микрочипов: влияние на обратную инженерию генных сетей». Биоинформатика. 23 (13): i282–8. Дои:10.1093 / биоинформатика / btm201. PMID 17646307.
- ^ Хохрайтер С., Клеверт Д.А., Обермайер К. (2006). «Новый метод суммирования данных уровня зонда affymetrix». Биоинформатика. 22 (8): 943–949. Дои:10.1093 / биоинформатика / btl033. PMID 16473874.
- ^ «Affycomp III: эталон для измерения экспрессии Affymetrix GeneChip».
- ^ Ши Л., Рид Л. Х., Джонс В. Д. и др. (2006). «Проект MicroArray Quality Control (MAQC) демонстрирует межплатформенную и внутриплатформенную воспроизводимость измерений экспрессии генов». Nat. Биотехнология. 24 (9): 1151–61. Дои:10.1038 / nbt1239. ЧВК 3272078. PMID 16964229.
- ^ Го Л., Лобенхофер Е.К., Ван С. и др. (2006). «Токсикогеномное исследование крыс показывает аналитическую согласованность на разных платформах микрочипов». Nat. Биотехнология. 24 (9): 1162–9. Дои:10.1038 / nbt1238. PMID 17061323. S2CID 8192240.
- ^ Джентльмен, Роберт; и другие. (2005). Решения для биоинформатики и вычислительной биологии с использованием R и Bioconductor. Нью-Йорк: Springer Science + Business Media. ISBN 978-0-387-29362-2.
- ^ Jaskowiak, Pablo A .; Кампелло, Рикардо J.G.B .; Коста, Иван Г. (2013). «Меры близости для кластеризации данных микрочипа экспрессии генов: методология проверки и сравнительный анализ». IEEE / ACM Transactions по вычислительной биологии и биоинформатике. 10 (4): 845–857. Дои:10.1109 / TCBB.2013.9. PMID 24334380. S2CID 760277.
- ^ а б c Ясковяк, Пабло А; Кампелло, Рикардо Дж.Б. Коста, Иван Г (2014). «О выборе подходящих расстояний для кластеризации данных по экспрессии генов». BMC Bioinformatics. 15 (Приложение 2): S2. Дои:10.1186 / 1471-2105-15-S2-S2. ЧВК 4072854. PMID 24564555.
- ^ а б de Souto, Marcilio C.P .; Коста, Иван Г .; de Araujo, Daniel S.A .; Людермир, Тереза Б .; Шлип, Александр (2008). «Кластеризация данных экспрессии гена рака: сравнительное исследование». BMC Bioinformatics. 9 (1): 497. Дои:10.1186/1471-2105-9-497. ЧВК 2632677. PMID 19038021.
- ^ http://www.biostat.ucsf.edu
- ^ "Системы изобретательности". Получено 2007-12-31.
- ^ "Геномика Ариадны: Pathway Studio". Архивировано из оригинал на 2007-12-30. Получено 2007-12-31.
- ^ «FunRich: функциональный анализ обогащения». Получено 2014-09-09.
- ^ [«Анализ значимости микрочипов». Получено 2007-12-31.]
- ^ «Программное обеспечение - Широкое». Получено 2007-12-31.
- ^ Субраманиан А., Тамайо П., Мутха В.К. и др. (2005). «Анализ обогащения набора генов: основанный на знаниях подход к интерпретации профилей экспрессии в масштабе всего генома». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 102 (43): 15545–50. Дои:10.1073 / pnas.0506580102. ЧВК 1239896. PMID 16199517.
- ^ «Биокарта - прокладывание жизненных путей». Получено 2007-12-31.
- ^ Vinayagam A, Hu Y, Kulkarni M, Roesel C, et al. (2013). «Структура анализа на основе белковых комплексов для наборов данных с высокой пропускной способностью. 6, RS5 (2013)». Sci. Сигнал. 6 (r5): rs5. Дои:10.1126 / scisignal.2003629. ЧВК 3756668. PMID 23443684.
- ^ "DBI Web". Архивировано из оригинал на 2007-07-05. Получено 2007-12-31.
- ^ "ОБЪЕМ". Получено 2007-12-31.
- ^ «РссГск». Получено 2008-10-15.
- ^ а б c d е ж грамм час я Чу, Г., Нарасимхан, Б., Тибширани, Р., Тушер, В. "SAM" Анализ значимости микрочипов "Руководство пользователя и технический документ". [1]
- ^ а б c d е ж грамм час я Zang, S .; Guo, R .; и другие. (2007). «Интеграция методов статистического вывода и новой меры контроля для повышения чувствительности и специфичности анализа данных в исследованиях профилирования экспрессии». Журнал биомедицинской информатики. 40 (5): 552–560. Дои:10.1016 / j.jbi.2007.01.002. PMID 17317331.
- ^ а б c d е ж грамм час я <Чжан, С. (2007). «Комплексная оценка SAM, R-пакета SAM и простая модификация для улучшения его характеристик». BMC Bioinformatics 8: 230.
- ^ а б c Тушер, В. Г .; Tibshirani, R .; и другие. (2001). «Анализ значимости микрочипов применительно к отклику на ионизирующее излучение» (PDF). Труды Национальной академии наук. 98 (9): 5116–5121. Bibcode:2001PNAS ... 98.5116G. Дои:10.1073 / pnas.091062498. ЧВК 33173. PMID 11309499.
- ^ Dinu, I.P .; JD; Мюллер, Т; Лю, Q; Adewale, AJ; Джангри, GS; Эйнеке, G; Фамульски, К.С.; Halloran, P; Ясуи, Ю. (2007). «Улучшение анализа набора генов данных микрочипов с помощью SAM-GS». BMC Bioinformatics. 8: 242. Дои:10.1186/1471-2105-8-242. ЧВК 1931607. PMID 17612399.
- ^ Джеффри, И. Х .; DG; Калхейн, AC. (2006). «Сравнение и оценка методов создания списков дифференциально экспрессируемых генов из данных микрочипа». BMC Bioinformatics. 7: 359. Дои:10.1186/1471-2105-7-359. ЧВК 1544358. PMID 16872483.
- ^ Ларссон, О. В. С; Тиммонс, Дж. А. (2005). «Соображения при использовании алгоритма анализа значимости микроматриц (SAM)». BMC Bioinformatics. 6: 129. Дои:10.1186/1471-2105-6-129. ЧВК 1173086. PMID 15921534.
- ^ Уилсон CL, Миллер CJ (2005). «Simpleaffy: пакет BioConductor для контроля качества Affymetrix и анализа данных». Биоинформатика. 21 (18): 3683–5. Дои:10.1093 / биоинформатика / bti605. PMID 16076888.
- ^ "Институт Дж. Крейга Вентера - Программное обеспечение". Получено 2008-01-01.
- ^ «Agilent | GeneSpring GX». Получено 2008-01-02.
- ^ "Ocimum Biosolutions | Genowiz". Архивировано из оригинал в 2009-11-24. Получено 2009-04-02.
внешняя ссылка
- ArrayExplorer - сравнивайте микрочипы бок о бок, чтобы найти тот, который лучше всего соответствует вашим исследовательским потребностям
- FARMS - Факторный анализ для надежного суммирования микрочипов, пакет R -программного обеспечения
- StatsArray - Online Microarray Analysis Services -программного обеспечения
- ArrayMining.net - веб-приложение для онлайн-анализа данных микрочипов -программного обеспечения
- FunRich - Выполнение анализа обогащения набора генов -программного обеспечения
- Сравнительный анализ транскриптомики в Справочный модуль по естественным наукам
- Инструкция по загрузке SAM
- GeneChip® Expression Analysis - основы анализа данных (от Affymetrix)
- Герцог data_analysis_fundamentals_manual