WikiDer > Проект человеческого микробиома
| Проект человеческого микробиома (HMP) | |
|---|---|
 | |
| Владелец | НАС Национальные институты здоровья |
| Создано | 2007 |
| Отключен | 2016 |
| Интернет сайт | hmpdacc |
В Проект человеческого микробиома (HMP) был США Национальные институты здоровья (NIH) исследовательская инициатива по улучшению понимания микробная флора участвует в здоровье и болезнях человека. Запущен в 2007 году,[1] первая фаза (HMP1) была сосредоточена на идентификации и характеристике микробной флоры человека. Вторая фаза, известная как Интегрированный проект микробиома человека (iHMP), была запущена в 2014 году с целью генерирования ресурсов для характеристики микробиом и выяснение роли микробов в состояниях здоровья и болезней. Программа получила финансирование в размере 170 миллионов долларов США. Общий фонд NIH с 2007 по 2016 гг.[2]
Важными компонентами HMP были независимые от культуры методы микробного сообщество характеристика, например метагеномика (который обеспечивает широкий генетический взгляд на отдельное микробное сообщество), а также обширные секвенирование всего генома (который обеспечивает «глубокий» генетический взгляд на определенные аспекты данного микробного сообщества, т.е. отдельных видов бактерий). Последний служил ссылкой геномные последовательности - 3000 таких последовательностей отдельных бактериальных изолирует в настоящее время запланированы - для сравнения при последующем метагеномном анализе. В рамках проекта также профинансировано глубокое секвенирование бактериального 16S рРНК последовательности, амплифицированные полимеразной цепной реакции от людей.[3]
Введение
До запуска HMP в популярных средствах массовой информации и научной литературе часто сообщалось, что в человеческом теле примерно в 10 раз больше микробных клеток и в 100 раз больше микробных генов, чем человеческих клеток; эта цифра была основана на оценках того, что микробиом человека включает около 100 триллионов бактериальных клеток, а у взрослого человека обычно около 10 триллионов человеческих клеток.[4] В 2014 г. Американская академия микробиологии опубликовал FAQ, в котором подчеркивается, что количество микробных клеток и количество человеческих клеток являются оценочными, и отмечалось, что недавние исследования пришли к новой оценке количества человеческих клеток примерно в 37 триллионов клеток, что означает, что соотношение микробных клеток к клеткам человека составляет примерно 3: 1.[4][5] В 2016 году другая группа опубликовала новую оценку соотношения примерно 1: 1 (1,3: 1, с «неопределенностью 25% и вариацией 53% по популяции стандартных самцов 70 кг»).[6][7]
Несмотря на ошеломляющее количество микробов внутри и на теле человека, мало что было известно об их роли в здоровье человека и болезнях. Многие организмы, составляющие микробиом, не выжили. культурный, идентифицированные или иным образом охарактеризованные. Однако организмы, которые, как считается, обнаруживаются в микробиоме человека, в целом могут быть отнесены к категории бактерии, Члены домен Археи, дрожжи, и одноклеточные эукариоты а также различные гельминт паразиты и вирусы, последние включают вирусы, которые заражают организмы клеточного микробиома (например, бактериофаги). Задача HMP - открыть и охарактеризовать микробиом человека, уделяя особое внимание участкам полости рта, кожи, влагалища, желудочно-кишечного тракта и дыхательных путей.
HMP будет решать некоторые из самых вдохновляющих, неприятных и фундаментальных научных вопросов сегодня. Важно отметить, что он также может разрушить искусственные барьеры между медицинской микробиологией и микробиологией окружающей среды. Есть надежда, что HMP не только определит новые способы определения состояния здоровья и предрасположенности к заболеваниям, но также определит параметры, необходимые для разработки, реализации и мониторинга стратегий преднамеренного манипулирования микробиотой человека, чтобы оптимизировать ее работу в контексте физиологии человека. .[8]
HMP был описан как «логическое концептуальное и экспериментальное расширение Проект "Геном человека"."[8] В 2007 году HMP был включен в Дорожную карту медицинских исследований NIH.[9] как один из Новые пути к открытиям. Организованная характеристика микробиома человека также проводится на международном уровне под эгидой Международного консорциума микробиома человека.[10] В Канадские институты медицинских исследованийчерез Институт инфекций и иммунитета CIHR возглавляет Канадскую микробиомную инициативу по разработке скоординированных и целенаправленных исследовательских усилий по анализу и характеристике микробов, колонизирующих человеческое тело, и их потенциального изменения во время хронических заболеваний.[11]
Учреждения-спонсоры
В HMP приняли участие многие исследовательские институты, в том числе Стэндфордский Университет, то Broad Institute, Университет Содружества Вирджинии, Вашингтонский университет, Северо-Восточный университет, Массачусетский технологический институт, то Медицинский колледж Бейлора, и многие другие. Вклады включали оценку данных, построение наборов данных эталонной последовательности, этические и юридические исследования, разработку технологий и многое другое.
Фаза первая (2007-2014)
HMP1 включал исследовательские работы многих организаций.[12] HMP1 поставил следующие цели:[13]
- Разработать эталонный набор последовательностей микробного генома и выполнить предварительную характеристику микробиома человека.
- Изучите связь между болезнью и изменениями в микробиоме человека.
- Разработка новых технологий и инструментов для вычислительного анализа
- Создать хранилище ресурсов
- Изучите этические, правовые и социальные последствия исследования микробиома человека.
Второй этап (2014-2016)
В 2014 году NIH запустил вторую фазу проекта, известную как Проект интегративного микробиома человека (iHMP). Целью iHMP было создание ресурсов для создания полной характеристики микробиома человека с упором на понимание присутствия микробиоты в состояниях здоровья и болезней.[14] Миссия проекта, как заявили NIH, заключалась в следующем:
В рамках iHMP будут созданы интегрированные продольные наборы данных биологических свойств как микробиома, так и хозяина из трех различных когортных исследований связанных с микробиомом состояний с использованием нескольких технологий «омики».[14]
Проект включал в себя три подпроекта, реализованных в нескольких учреждениях. Включенные методы исследования 16S рРНК генное профилирование, целое метагеном секвенирование дробовика, секвенирование всего генома, метатранскриптомика, метаболомика/липидомика, и иммунопротеомика. Основные выводы iHMP были опубликованы в 2019 году.[15]
Беременность и преждевременные роды
Команда Консорциума влагалищного микробиома в Университете Содружества Вирджинии провела исследование проекта «Беременность и преждевременные роды» с целью понять, как микробиом изменяется во время гестационного периода и влияет на микробиом новорожденного. Проект также был посвящен роли микробиома в возникновении преждевременных родов, которые, по мнению CDC, составляют почти 10% всех рождений[16] и составляет вторую по значимости причину неонатальной смерти.[17] Проект получил финансирование NIH в размере 7,44 миллиона долларов.[18]
Начало воспалительного заболевания кишечника (ВЗК)
Команда Multi'omics Data по воспалительному заболеванию кишечника (IBDMDB) представляла собой группу исследователей из нескольких учреждений, сосредоточенных на понимании того, как микробиом кишечника продольно изменяется у взрослых и детей, страдающих IBD. ВЗК - воспалительный аутоиммунное заболевание что проявляется как болезнь Крона или язвенный колит и затрагивает около миллиона американцев.[19] Участниками исследования были когорты из Массачусетская больница общего профиля, Университетская больница Эмори/Детская больница Цинциннати, и Седарс-Синайский медицинский центр.[20]
Начало диабета 2 типа (СД2)
Исследователи из Стэнфордского университета и Джексонская лаборатория геномной медицины работали вместе, чтобы выполнить продольный анализ биологических процессов, которые происходят в микробиоме пациентов с риском Диабет 2 типа. СД2 поражает почти 20 миллионов американцев, по крайней мере, у 79 миллионов пациентов с преддиабетом.[21] и частично характеризуется заметными сдвигами в микробиоме по сравнению со здоровыми людьми. Проект направлен на выявление молекул и сигнальных путей, которые играют роль в этиология болезни.[22]
Достижения
Воздействие HMP на сегодняшний день может быть частично оценено путем изучения исследований, спонсируемых HMP. С июня 2009 г. по конец 2017 г. на веб-сайте HMP было размещено более 650 рецензируемых публикаций, которые были процитированы более 70 000 раз.[23] На данный момент веб-сайт был заархивирован и больше не обновляется, хотя наборы данных по-прежнему доступны.[24]
Основные категории работ, финансируемых HMP, включали:
- Разработка новых систем баз данных, обеспечивающих эффективную организацию, хранение, доступ, поиск и аннотирование огромных объемов данных. К ним относятся IMG, Интегрированные микробные геномы база данных и система сравнительного анализа;[25] IMG / M, родственная система, которая объединяет наборы данных метагенома с изолированными геномами микробов из системы IMG;[26] CharProtDB, база данных экспериментально охарактеризованных аннотаций белков;[27] и База данных Genomes OnLine (GOLD) для мониторинга состояния геномных и метагеномных проектов во всем мире и связанных с ними метаданных.[28]
- Разработка инструментов для сравнительного анализа, которые облегчают распознавание общих закономерностей, основных тем и тенденций в сложных наборах данных. К ним относятся RAPSearch2, быстрый и эффективный с точки зрения памяти инструмент поиска сходства белков для данных секвенирования следующего поколения;[29] Boulder ALignment Editor (ALE), веб-инструмент для выравнивания РНК;[30] WebMGA, настраиваемый веб-сервер для быстрого анализа метагеномной последовательности;[31] и DNACLUST, инструмент для точной и эффективной кластеризации генов филогенетических маркеров.[32]
- Разработка новых методов и систем для сборки массивных наборов данных последовательности. Нет единого алгоритма сборки, решающего все известные проблемы сборки коротких последовательностей,[33] поэтому программы сборки нового поколения, такие как AMOS[34] имеют модульную конструкцию и предлагают широкий набор инструментов для сборки. Новые алгоритмы были разработаны для улучшения качества и полезности черновых последовательностей генома.[35]
- Сборка каталога секвенированных эталонных геномов чистых бактериальных штаммов из нескольких участков тела, с которыми можно сравнивать метагеномные результаты. Первоначальная цель - 600 геномов - далеко превзойдена; в настоящее время цель состоит в том, чтобы в этот справочный каталог было включено 3000 геномов, секвенированных, по крайней мере, до стадии высококачественного черновика. По состоянию на март 2012 г.[Обновить]Каталогизировано 742 генома.[36]
- Создание Центра анализа и координации данных (DACC),[37] который служит центральным хранилищем всех данных HMP.
- Различные исследования, изучающие правовые и этические вопросы, связанные с исследованиями секвенирования всего генома.[38][39][40][41]
Разработки, финансируемые HMP, включали:
- Новые методы прогнозирования для выявления активных сайтов связывания факторов транскрипции.[42]
- Идентификация на основе биоинформатических данных широко распространенного предшественника электронного носителя, продуцируемого рибосомами.[43]
- Покадровые "движущиеся изображения" человеческого микробиома.[44]
- Выявление уникальных приспособлений, принятых сегментированными нитчатыми бактериями (SFB) в их роли кишечных комменсалов.[45] SFB важны с медицинской точки зрения, потому что они стимулируют Т-хелпер 17 клеток, считается, что играет ключевую роль в аутоиммунное заболевание.
- Выявление факторов, различающих микробиоту здорового и больного кишечника.[46]
- Определение до сих пор непризнанной доминирующей роли Веррукомикробия в почвенных бактериальных сообществах.[47]
- Выявление факторов, определяющих потенциал вирулентности Gardnerella vaginalis напряжения в вагиноз.[48]
- Выявление связи между микробиотой полости рта и атеросклерозом.[49]
- Демонстрация того, что патогенные виды Neisseria участвует в менингит, сепсис, и венерическая болезнь обмениваются факторами вирулентности с комменсальными видами.[50]
Вехи
Создана справочная база данных
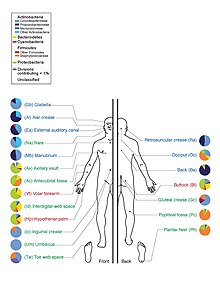
13 июня 2012 года главный вехой HMP был объявлен Национальные институты здравоохранения США директор Фрэнсис Коллинз.[51] Анонс сопровождался серией скоординированных статей, опубликованных в Природа[52][53] и несколько журналов, включая Публичная научная библиотека (PLoS) в тот же день.[54][55][56][57][58][59] Картируя нормальный микробный состав здоровых людей с помощью методов секвенирования генома, исследователи HMP создали справочную базу данных и границы нормальных микробных вариаций у людей.[60]
У 242 здоровых добровольцев из США было собрано более 5000 образцов тканей с 15 (мужчин) до 18 (женщин) участков тела, таких как рот, нос, кожа, нижняя часть кишечника (стул) и влагалище. Вся ДНК, человеческая и микробная, была проанализирована с помощью машин для секвенирования ДНК. Данные микробного генома были извлечены путем идентификации бактериальной специфической рибосомной РНК, 16S рРНК. Исследователи подсчитали, что более 10000 видов микробов населяют экосистему человека, и они идентифицировали 81-99% роды. Помимо создания справочной базы данных микробиома человека, проект HMP также обнаружил несколько «сюрпризов», в том числе:
- Микробы вносят больше генов, ответственных за выживание человека, чем собственных генов человека. Подсчитано, что бактериальные гены, кодирующие белок, в 360 раз более распространены, чем гены человека.
- Метаболическая активность микробов; например, переваривание жиров; не всегда представлены одними и теми же видами бактерий. Наличие занятий, кажется, имеет большее значение.
- Компоненты микробиома человека меняются со временем в зависимости от болезненного состояния пациента и приема лекарств. Однако микробиом в конечном итоге возвращается в состояние равновесия, хотя состав бактерий изменился.
Клиническое применение
Среди первых клинических приложений, использующих данные HMP, как сообщается в нескольких статьях PLoS, исследователи обнаружили переход к меньшему видовому разнообразию в вагинальный микробиом беременных женщин, готовящихся к родам, и высокой вирусной ДНК-нагрузкой в носовом микробиоме детей с необъяснимой лихорадкой. Другие исследования с использованием данных и методов HMP включают роль микробиома в различных заболеваниях пищеварительного тракта, кожи, репродуктивных органов и детских расстройствах.[51]
Фармацевтическое применение
Фармацевтические микробиологи рассмотрели значение данных HMP в отношении наличия / отсутствия «нежелательных» микроорганизмов в нестерильных фармацевтических продуктах и в отношении мониторинга микроорганизмов в контролируемой среде, в которой производятся продукты. Последнее также имеет значение для выбора среды и исследований эффективности дезинфицирующих средств.[61]
Смотрите также
использованная литература
- ^ "Проект" Микробиом человека: разнообразие человеческих микробов больше, чем предполагалось ". ScienceDaily. Получено 8 марта 2012.
- ^ "Проект микробиома человека - Главная | Общий фонд NIH". commonfund.nih.gov. Получено 2018-04-15.
- ^ «Проект человеческого микробиома». Общий фонд NIH. Получено 8 марта 2012.
- ^ а б Американская академия микробиологии Часто задаваемые вопросы: микробиом человека В архиве 2016-12-31 в Wayback Machine Январь 2014
- ^ Джуда Л. Рознер для журнала Microbe Magazine, февраль 2014 г. У человека в десять раз больше микробных клеток, чем клеток тела?
- ^ Элисон Эбботт для Nature News. 8 января 2016 г. Ученые опровергают миф о том, что в нашем организме больше бактерий, чем клеток человека
- ^ Отправитель Р., Фукс С., Майло Р. (январь 2016 г.). «Действительно ли нас значительно меньше численности? Возвращаясь к соотношению бактериальных клеток и клеток-хозяев у людей». Ячейка. 164 (3): 337–40. Дои:10.1016 / j.cell.2016.01.013. PMID 26824647.
- ^ а б Тернбо П.Дж., Лей Р.Э., Хамади М., Фрейзер-Лиггетт С.М., Найт Р., Гордон Д.И. (октябрь 2007 г.). «Проект микробиома человека». Природа. 449 (7164): 804–10. Bibcode:2007Натура.449..804Т. Дои:10.1038 / природа06244. ЧВК 3709439. PMID 17943116.
- ^ «О дорожной карте NIH». Общий фонд NIH. Архивировано из оригинал 17 февраля 2013 г.. Получено 8 марта 2012.
- ^ «Международный консорциум микробиома человека». Получено 8 марта 2012.
- ^ «Канадская инициатива по микробиому». Канадские институты медицинских исследований. Получено 8 марта 2012.
- ^ «Проект микробиома человека / финансируемое исследование». Общий фонд NIH. Получено 8 марта 2012.
- ^ «Проект по микробиому человека / программные инициативы». Общий фонд NIH. Получено 8 марта 2012.
- ^ а б "Проект NIH Human Microbiome - О микробиоме человека". hmpdacc.org. Получено 2018-03-30.
- ^ Группа анализа портфеля микробиома человека NIH (2019). «Обзор 10-летней деятельности по исследованию микробиома человека в Национальных институтах здравоохранения США, 2007-2016 финансовые годы». Микробиом. 7 (1): 31. Дои:10.1186 / s40168-019-0620-у. ISSN 2049-2618. ЧВК 6391833. PMID 30808411.
- ^ Ферре С., Каллаган В., Олсон С., Шарма А., Барфилд В. (ноябрь 2016 г.). «Влияние возраста матери и возрастной частоты преждевременных родов на общую частоту преждевременных родов - США, 2007 и 2014 годы». MMWR. Еженедельный отчет о заболеваемости и смертности. 65 (43): 1181–1184. Дои:10,15585 / ммwr.mm6543a1. PMID 27811841.
- ^ "Младенческая смертность | Здоровье матери и ребенка | Репродуктивное здоровье | CDC". www.cdc.gov. 2018-01-02. Получено 2018-04-03.
- ^ Консорциум, VCU, Vaginal Microbiome. «Консорциум вагинального микробиома». vmc.vcu.edu. Получено 2018-04-05.
- ^ «CDC - Эпидемиология IBD - воспалительного заболевания кишечника». www.cdc.gov. Архивировано из оригинал на 2017-02-23. Получено 2018-04-15.
- ^ "Команда". ibdmdb.org. Получено 2018-04-05.
- ^ "Национальный статистический отчет по диабету | Данные и статистика | Диабет | CDC". www.cdc.gov. 2018-03-09. Получено 2018-04-15.
- ^ «Интегрированное профилирование личного омика | Комплексное профилирование личного омика | Стэнфордская медицина». med.stanford.edu. Получено 2018-04-05.
- ^ "Проект микробиома человека - Главная | Общий фонд NIH". commonfund.nih.gov. Получено 2019-04-18.
- ^ "Портал данных проекта" Микробиом человека ". portal.hmpdacc.org. Получено 2019-04-18.
- ^ Марковиц В.М., Чен И.М., Паланиаппан К., Чу К., Сзето Э., Гречкин Ю., Ратнер А., Джейкоб Б., Хуанг Дж., Уильямс П., Хантеманн М., Андерсон И., Мавроматис К., Иванова Н. Н., Кирпидес NC (январь 2012 г.). «IMG: интегрированная база данных микробных геномов и система сравнительного анализа». Исследования нуклеиновых кислот. 40 (Выпуск базы данных): D115–22. Дои:10.1093 / nar / gkr1044. ЧВК 3245086. PMID 22194640.
- ^ Марковиц В.М., Чен И.М., Чу К., Сзето Э., Паланиаппан К., Гречкин Ю., Ратнер А., Джейкоб Б., Пати А., Хантеманн М., Лиолиос К., Пагани И., Андерсон И., Мавроматис К., Иванова Н. Н., Кирпидес NC (январь 2012 г. ). «IMG / M: интегрированная система управления данными метагенома и сравнительного анализа». Исследования нуклеиновых кислот. 40 (Выпуск базы данных): D123–9. Дои:10.1093 / nar / gkr975. ЧВК 3245048. PMID 22086953.
- ^ Мадупу Р., Рихтер А., Додсон Р.Дж., Бринкач Л., Харкинс Д., Дуркин С., Шривастава С., Саттон Г., Хафт Д. (январь 2012 г.). «CharProtDB: база данных экспериментально охарактеризованных аннотаций белков». Исследования нуклеиновых кислот. 40 (Выпуск базы данных): D237–41. Дои:10.1093 / нар / gkr1133. ЧВК 3245046. PMID 22140108.
- ^ Пагани И., Лиолиос К., Янссон Дж., Чен И.М., Смирнова Т., Носрат Б., Марковиц В.М., Кирпидес, Северная Каролина (январь 2012 г.). «База данных Genomes OnLine (GOLD) v.4: состояние геномных и метагеномных проектов и связанных с ними метаданных». Исследования нуклеиновых кислот. 40 (Выпуск базы данных): D571–9. Дои:10.1093 / nar / gkr1100. ЧВК 3245063. PMID 22135293.
- ^ Чжао Й, Тан Х, Е Й (январь 2012 г.). «RAPSearch2: быстрый и эффективный с точки зрения памяти инструмент поиска сходства белков для данных секвенирования следующего поколения». Биоинформатика. 28 (1): 125–6. Дои:10.1093 / биоинформатика / btr595. ЧВК 3244761. PMID 22039206.
- ^ Stombaugh J, Widmann J, McDonald D, Knight R (июнь 2011 г.). "Boulder ALignment Editor (ALE): веб-инструмент для выравнивания РНК". Биоинформатика. 27 (12): 1706–7. Дои:10.1093 / биоинформатика / btr258. ЧВК 3106197. PMID 21546392.
- ^ Ву С., Чжу З., Фу Л., Ню Б., Ли В. (сентябрь 2011 г.). «WebMGA: настраиваемый веб-сервер для быстрого анализа метагеномной последовательности». BMC Genomics. 12: 444. Дои:10.1186/1471-2164-12-444. ЧВК 3180703. PMID 21899761.
- ^ Годси М., Лю Б., Поп М. (июнь 2011 г.). «DNACLUST: точная и эффективная кластеризация генов филогенетических маркеров». BMC Bioinformatics. 12: 271. Дои:10.1186/1471-2105-12-271. ЧВК 3213679. PMID 21718538.
- ^ Яо Г., Е Л., Гао Х, Минкс П., Уоррен В. К., Вайншток Г. М. (январь 2012 г.). «График соответствия сборок последовательностей следующего поколения». Биоинформатика. 28 (1): 13–6. Дои:10.1093 / биоинформатика / btr588. ЧВК 3244760. PMID 22025481.
- ^ Treangen TJ, Sommer DD, Angly FE, Koren S, Pop M (март 2011 г.). Сборка последовательностей нового поколения с AMOS. Текущие протоколы в биоинформатике. Глава 11. С. Раздел 11.8. Дои:10.1002 / 0471250953.bi1108s33. ISBN 978-0471250951. ЧВК 3072823. PMID 21400694.
- ^ Корен С., Миллер-младший, Валенц Б. П., Саттон Дж. (Сентябрь 2010 г.). «Алгоритм автоматического закрытия при сборке». BMC Bioinformatics. 11: 457. Дои:10.1186/1471-2105-11-457. ЧВК 2945939. PMID 20831800.
- ^ "Проект по микробиому человека / Справочные данные по геномам". Центр анализа и координации данных (DACC) для Национальных институтов здравоохранения (NIH). Получено 8 марта 2012.
- ^ «Центр анализа и координации данных (DACC)». Общий фонд национальных институтов здравоохранения (NIH). Получено 11 марта 2012.
- ^ Шваб А.П., Франк Л., Глигоров Н. (ноябрь 2011 г.). «Говорить о конфиденциальности, имея в виду конфиденциальность». Американский журнал биоэтики. 11 (11): 44–5. Дои:10.1080/15265161.2011.608243. PMID 22047127. S2CID 34313782.
- ^ Родос Р., Аззуни Дж., Баумрин С.Б., Бенков К., Блазер М.Дж., Бреннер Б., Даубен Дж. В., Эрл В. Дж., Франк Л., Глигоров Н., Гольдфарб Дж., Хиршхорн К., Хиршхорн Р., Хольцман И., Индик Д., Джабс Е. В., Лаки Д. П. , Морос Д.А., Филпотт С., Родс М.Э., Ричардсон Л.Д., Сакс Х.С., Шваб А., Сперлинг Р., Труско Б., Цвейг А. (ноябрь 2011 г.). «Минимальный риск: предложение по новой категории исследовательского риска». Американский журнал биоэтики. 11 (11): 1–7. Дои:10.1080/15265161.2011.615588. PMID 22047112. S2CID 205859554.
- ^ Макгуайр А.Л., Лупски-младший (май 2010 г.). «Персональное исследование генома: что нужно сказать участнику?». Тенденции в генетике. 26 (5): 199–201. Дои:10.1016 / j.tig.2009.12.007. ЧВК 2868334. PMID 20381895.
- ^ Шарп Р.Р., Ачкар Дж. П., Бринич М.А., Фаррелл Р.М. (апрель 2009 г.). «Помощь пациентам сделать осознанный выбор в отношении пробиотиков: необходимость исследования». Американский журнал гастроэнтерологии. 104 (4): 809–13. Дои:10.1038 / ajg.2008.68. ЧВК 2746707. PMID 19343022.
- ^ Cuellar-Partida G, Buske FA, McLeay RC, Whitington T, Noble WS, Bailey TL (январь 2012 г.). «Эпигенетические приоры для идентификации сайтов связывания активных транскрипционных факторов». Биоинформатика. 28 (1): 56–62. Дои:10.1093 / биоинформатика / btr614. ЧВК 3244768. PMID 22072382.
- ^ Haft DH (январь 2011 г.). «Биоинформатические доказательства широко распространенного предшественника электронного носителя, продуцируемого рибосомами, его белков созревания и его никотинопротеиновых редокс-партнеров». BMC Genomics. 12: 21. Дои:10.1186/1471-2164-12-21. ЧВК 3023750. PMID 21223593.
- ^ Капорасо Дж. Г., Лаубер К. Л., Костелло Е. К., Берг-Лайонс Д., Гонсалес А., Стомбо Дж., Найтс Д., Гайер П., Равель Дж., Фирер Н., Гордон Дж. И., Рыцарь Р. (2011). «Движущиеся картинки микробиома человека». Геномная биология. 12 (5): R50. Дои:10.1186 / gb-2011-12-5-r50. ЧВК 3271711. PMID 21624126.
- ^ Щеснак А., Сегата Н., Цинь Х, Геверс Д., Петросино Дж. Ф., Хаттенхауэр С., Литтман Д. Р., Иванов И. И. (сентябрь 2011 г.). «Геном сегментированных нитчатых бактерий, индуцирующих клетки th17, обнаруживает обширную ауксотрофию и адаптацию к кишечной среде». Клеточный хозяин и микроб. 10 (3): 260–72. Дои:10.1016 / j.chom.2011.08.005. ЧВК 3209701. PMID 21925113.
- ^ Баллал С.А., Галлини КА, Сегата Н., Хаттенхауэр С., Гарретт WS (апрель 2011 г.). «Симбиотические факторы микробиоты хозяина и кишечника: уроки воспалительного заболевания кишечника и успешные симбионты». Клеточная микробиология. 13 (4): 508–17. Дои:10.1111 / j.1462-5822.2011.01572.x. PMID 21314883. S2CID 205529660.
- ^ Bergmann GT, Bates ST, Eilers KG, Lauber CL, Caporaso JG, Walters WA, Knight R, Fierer N (июль 2011 г.). «Недооцененное доминирование Verrucomicrobia в сообществах почвенных бактерий». Биология и биохимия почвы. 43 (7): 1450–1455. Дои:10.1016 / j.soilbio.2011.03.012. ЧВК 3260529. PMID 22267877.
- ^ Йоман С.Дж., Йилдирим С., Томас С.М., Дуркин А.С., Торральба М., Саттон Дж., Бухай С.Дж., Динг Й., Дуган-Роша С.П., Музны Д.М., Цинь Икс, Гиббс Р.А., Ли С.Р., Стампф Р., Уайт Б.А., Горец С.К., Нельсон К.Е., Уилсон Б.А. (август 2010 г.). Ли В. (ред.). «Сравнительная геномика штаммов Gardnerella vaginalis показывает существенные различия в метаболическом и вирулентном потенциале». PLOS ONE. 5 (8): e12411. Bibcode:2010PLoSO ... 512411Y. Дои:10.1371 / journal.pone.0012411. ЧВК 2928729. PMID 20865041.
- ^ Корен О, Спор А, Фелин Дж., Фак Ф., Стомбо Дж., Тремароли В., Бер Си Джей, Найт Р., Фагерберг Б., Лей Р. Э., Бэкхед Ф. (март 2011 г.). «Микробиота полости рта, кишечника и бляшек у пациентов с атеросклерозом». Труды Национальной академии наук Соединенных Штатов Америки. 108 Дополнение 1 (Дополнение_1): 4592–8. Bibcode:2011ПНАС..108.4592К. Дои:10.1073 / pnas.1011383107. ЧВК 3063583. PMID 20937873.
- ^ Marri PR, Paniscus M, Weyand NJ, Rendón MA, Calton CM, Hernández DR, Higashi DL, Sodergren E, Weinstock GM, Rounsley SD, So M (июль 2010 г.). Ахмед Н. (ред.). «Секвенирование генома показывает широко распространенный обмен генами вирулентности среди человеческих видов Neisseria». PLOS ONE. 5 (7): e11835. Bibcode:2010PLoSO ... 511835M. Дои:10.1371 / journal.pone.0011835. ЧВК 2911385. PMID 20676376.
- ^ а б «Проект NIH Human Microbiome Project определяет нормальный бактериальный состав тела». Новости NIH. 13 июня 2012 г.
- ^ Консорциум проекта микробиома человека (июнь 2012 г.). «Основа для исследования микробиома человека». Природа. 486 (7402): 215–21. Bibcode:2012Натура 486..215т. Дои:10.1038 / природа11209. ЧВК 3377744. PMID 22699610.
- ^ Консорциум проекта микробиома человека (июнь 2012 г.). «Структура, функции и разнообразие здорового микробиома человека». Природа. 486 (7402): 207–14. Bibcode:2012Натура 486..207Т. Дои:10.1038 / природа11234. ЧВК 3564958. PMID 22699609.
- ^ Фауст К., Сатирапонгсасути Дж. Ф., Изард Дж., Сегата Н., Геверс Д., Раес Дж., Хаттенхауэр С. (2012). «Взаимосвязи микробного сосуществования в микробиоме человека». PLOS вычислительная биология. 8 (7): e1002606. Bibcode:2012PLSCB ... 8E2606F. Дои:10.1371 / journal.pcbi.1002606. ЧВК 3395616. PMID 22807668.
- ^ Абубукер С., Сегата Н., Голл Дж., Шуберт А.М., Изард Дж., Кантарел Б.Л., Родригес-Мюллер Б., Цукер Дж., Тиагараджан М., Хенриссат Б., Уайт О, Келли С.Т., Мете Б., Шлосс П.Д., Геверс Д., Митрева М., Хаттенхауэр C (2012). «Метаболическая реконструкция метагеномных данных и ее применение к микробиому человека». PLOS вычислительная биология. 8 (6): e1002358. Bibcode:2012PLSCB ... 8E2358A. Дои:10.1371 / journal.pcbi.1002358. ЧВК 3374609. PMID 22719234.
- ^ Сегата Н., Хааке С.К., Маннон П., Лимон К.П., Уолдрон Л., Геверс Д., Хаттенхауэр С., Изард Дж. (Июнь 2012 г.). «Состав бактериального микробиома пищеварительного тракта взрослого человека на основе семи образцов поверхности рта, миндалин, горла и стула». Геномная биология. 13 (6): R42. Дои:10.1186 / gb-2012-13-6-r42. ЧВК 3446314. PMID 22698087.
- ^ Cantarel BL, Lombard V, Henrissat B (2012). «Использование сложных углеводов здоровым микробиомом человека». PLOS ONE. 7 (6): e28742. Bibcode:2012PLoSO ... 728742C. Дои:10.1371 / journal.pone.0028742. ЧВК 3374616. PMID 22719820.
- ^ Ву Ю.В., Ро М., Доак Т.Г., Йе Й. (август 2012 г.). «Спирохеты полости рта, причастные к стоматологическим заболеваниям, широко распространены у нормальных людей и несут чрезвычайно разнообразные кассеты интегронных генов». Прикладная и экологическая микробиология. 78 (15): 5288–96. Дои:10.1128 / AEM.00564-12. ЧВК 3416431. PMID 22635997.
- ^ «Коллекции PLOS: коллекции статей, опубликованные Публичной научной библиотекой». collections.plos.org. Получено 2018-04-15.
- ^ Резюме рукописи
- ^ Уайлдер К., Сандл Т., Саттон С. (июнь 2013 г.). «Влияние человеческого микробиома на фармацевтическую микробиологию». Американский фармацевтический обзор.
внешние ссылки
- Проект человеческого микробиома
- Центр анализа и координации данных
- Канадская инициатива по микробиому CIHR
- Международный консорциум микробиома человека
- 2006, Краткое изложение исследования микробиома толстой кишки (фактическое исследование: Gill et al., 2006)
- Оливия Джадсон Microbes ‘R’ Us Газета "Нью-Йорк Таймс 22 июля 2009 г.
- Джина Колата Хорошее здоровье? Поблагодарите свои 100 триллионов бактерий Газета "Нью-Йорк Таймс 13 июня 2012 г.