WikiDer > Мониторинг выбранных реакций
Мониторинг выбранных реакций (SRM) - метод, используемый в тандемная масс-спектрометрия в котором ион определенной массы выбирается на первом этапе тандема масс-спектрометр и ионный продукт реакции фрагментации иона-предшественника выбирается на втором этапе масс-спектрометра для обнаружения.[1]
Варианты
Общий случай SRM можно представить как
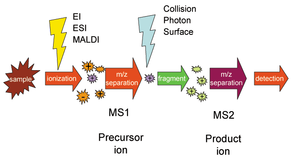
где ион-предшественник ABCD+ выбирается на первом этапе масс-спектрометрии (MS1), диссоциирует на молекулу AB и ион продукта CD+, а последний выбирается на втором этапе масс-спектрометрии (MS2) и детектируется. Ионная пара предшественника и продукта называется «переходом» SRM. [2]
Последовательный мониторинг реакции (CRM) представляет собой последовательное применение трех или более стадий масс-спектрометрии к SRM, представленное в простом случае как
где ABCD+ выбирается MS1, диссоциирует на молекулу AB и ион CD+.[3] Ион выбирается на второй стадии масс-спектрометрии MS2, затем подвергается дальнейшей фрагментации с образованием иона D.+ который выбирается на третьем этапе масс-спектрометрии MS3 и обнаруживается.
Мониторинг множественных реакций (MRM) представляет собой применение мониторинга выбранной реакции к множественным ионам продуктов из одного или нескольких ионов-предшественников,[3][4] Например
где ABCD+ выбирается MS1 и диссоциирует двумя путями, образуя либо AB+ или компакт-диск+. Ионы последовательно выбираются MS2 и детектируются. Мониторинг параллельных реакций (PRM) - это применение SRM с параллельным обнаружением всех переходов в одном анализе с использованием масс-спектрометра высокого разрешения.[5]
Протеомика
SRM можно использовать для целевых количественная протеомика к масс-спектрометрии.[6] Следующий ионизация в, например, электроспрей источник, а пептид прекурсор сначала выделяется для получения существенного ион популяция в основном предполагаемых видов. Тогда это население фрагментированный чтобы получить ионы продукта, содержание сигналов которых указывает на содержание пептида в образце. Этот эксперимент можно провести на тройные квадрупольные масс-спектрометры, где разрешающая масса Q1 изолирует предшественник, q2 действует как столкновительная ячейка, а разрешающая масса Q3 проходит через ионы продукта, которые обнаруживаются при выходе из последнего квадруполь по электронный умножитель. Пару прекурсор / продукт часто называют переход. Большая работа направлена на то, чтобы выбрать переходы с максимальной специфичностью.
С помощью изотопная маркировка с тяжелыми метками (например, D, 13C, или же 15N) пептиды к сложной матрице как стандарты концентрации, SRM можно использовать для построения калибровочная кривая который может обеспечить абсолютную количественную оценку (т. е. количество копий на клетка) нативного легкого пептида и, соответственно, его родительского белок.[2]
SRM использовался для идентификации белков, кодируемых генами дикого типа и мутантными генами (мутантные белки) и количественно определить их абсолютное количество копий в опухолях и биологических жидкостях, тем самым ответив на основные вопросы об абсолютном количестве копий белков в одной клетке, что будет иметь важное значение при цифровом моделировании клеток млекопитающих и человеческого тела, а также об относительных уровнях генетически аномальные белки в опухолях и доказывают свою полезность для диагностических приложений.[7][8] SRM также использовался в качестве метода запуска сканирования полного ионного продукта пептидов для а) подтверждения специфичности перехода SRM или б) обнаружения специфических посттрансляционные модификации которые ниже предела обнаружения стандартных анализов МС.[9] В 2017 году SRM была разработана как высокочувствительная и воспроизводимая платформа для целевого обнаружения белков на основе масс-спектрометрии (названная «SAFE-SRM»), и было продемонстрировано, что новый конвейер на основе SRM имеет основные преимущества в приложениях клинической протеомики. по сравнению с традиционными конвейерами SRM, и он продемонстрировал значительно улучшенные диагностические характеристики по сравнению с методами диагностики белковых биомаркеров на основе антител, такими как ELISA.[10]
Смотрите также
Рекомендации
- ^ Э. де Хоффманн (1996). «Тандемная масс-спектрометрия: праймер» (PDF). Журнал масс-спектрометрии. 31 (2): 129–137. Дои:10.1002 / (SICI) 1096-9888 (199602) 31: 2 <129 :: AID-JMS305> 3.0.CO; 2-T.
- ^ а б Ланге, Винзенц; Пикотти, Паола; Домон, Бруно; Эберсольд, Руеди (2008). «Выборочный мониторинг реакций для количественной протеомики: учебное пособие». Молекулярная системная биология. 4: 222. Дои:10.1038 / msb.2008.61. ISSN 1744-4292. ЧВК 2583086. PMID 18854821.
- ^ а б Мюррей, Кермит К .; Бойд, Роберт К .; Eberlin, Marcos N .; Лэнгли, Дж. Джон; Ли, Лян; Найто, Ясухидэ (2013). «Определения терминов, относящихся к масс-спектрометрии (Рекомендации ИЮПАК 2013 г.)». Чистая и прикладная химия. 85 (7): 1515–1609. Дои:10.1351 / PAC-REC-06-04-06. ISSN 1365-3075.
- ^ Кондрат, Р. У .; McClusky, G.A .; Кук, Р. Г. (1978). «Мониторинг множественных реакций в масс-спектрометрии / масс-спектрометрии для прямого анализа сложных смесей». Аналитическая химия. 50 (14): 2017–2021. Дои:10.1021 / ac50036a020. ISSN 0003-2700.
- ^ Peterson, A.C .; Russell, J.D .; Бейли, Д. Дж .; Westphall, M. S .; Кун, Дж. Дж. (2012). «Мониторинг параллельных реакций для количественной целевой протеомики с высоким разрешением и высокой точностью масс». Молекулярная и клеточная протеомика. 11 (11): 1475–1488. Дои:10.1074 / mcp.O112.020131. ISSN 1535-9476. ЧВК 3494192. PMID 22865924.
- ^ Пикотти, Паола; Эберсольд, Руеди (2012). «Протеомика, основанная на мониторинге избранных реакций: рабочие процессы, потенциал, подводные камни и будущие направления». Методы природы. 9 (6): 555–566. Дои:10.1038 / nmeth.2015. ISSN 1548-7091. PMID 22669653.
- ^ Ван Кью, Чаркади Р., Ву Дж и др. (Февраль 2011 г.). «Мутантные белки как биомаркеры рака». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 108 (6): 2444–9. Bibcode:2011PNAS..108.2444W. Дои:10.1073 / pnas.1019203108. ЧВК 3038743. PMID 21248225.
- ^ «Масс-спектрометрия с мониторингом выбранной реакции для определения абсолютного белка». Журнал визуализированных экспериментов.
- ^ Анвин Р.Д., Гриффитс Дж. Г. и др. (Август 2005 г.). «Мониторинг множественных реакций для выявления участков фосфорилирования белков с высокой чувствительностью». Молекулярная и клеточная протеомика. 4 (8): 1134–44. Дои:10.1074 / mcp.M500113-MCP200. PMID 15923565.
- ^ Ван Кью, Чжан М., Томита Т. и др. (Декабрь 2017 г.). «Выбранный подход к мониторингу реакций для проверки пептидных биомаркеров». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 114 (51): 13519–13524. Дои:10.1073 / пнас.1712731114. ЧВК 5754789. PMID 29203663.