WikiDer > Секвенирование ДНК
| Часть серия на |
| Генетика |
|---|
 |
| Ключевые компоненты |
| История и темы |
| Исследование |
| Персонализированная медицина |
| Персонализированная медицина |
Секвенирование ДНК это процесс определения последовательность нуклеиновой кислоты - получатель чего-то нуклеотиды в ДНК. Он включает любой метод или технологию, которые используются для определения порядка четырех оснований: аденин, гуанин, цитозин, и тимин. Появление методов быстрого секвенирования ДНК значительно ускорило биологические и медицинские исследования и открытия.[1][2]
Знание Последовательности ДНК стала незаменимой для фундаментальных биологических исследований и во многих прикладных областях, таких как медицинский диагноз, биотехнология, судебная биология, вирусология и биологические систематика. Сравнение здоровых и мутировавших последовательностей ДНК может диагностировать различные заболевания, включая различные виды рака,[3] охарактеризовать репертуар антител,[4] и может использоваться для руководства лечением пациента.[5] Наличие быстрого способа секвенирования ДНК позволяет оказывать более быструю и индивидуализированную медицинскую помощь, а также идентифицировать и каталогизировать больше организмов.[4]
Высокая скорость секвенирования, достигаемая с помощью современных технологий секвенирования ДНК, сыграла важную роль в секвенировании полных последовательностей ДНК или геномы, многочисленных типов и видов жизни, в том числе человеческий геном и другие полные последовательности ДНК многих видов животных, растений и микробов.
Первые последовательности ДНК были получены в начале 1970-х годов академическими исследователями с использованием трудоемких методов, основанных на двумерная хроматография. После разработки флуоресценция-основные методы секвенирования с Секвенатор ДНК,[6] Секвенирование ДНК стало проще и на порядки быстрее.[7]
Приложения
Секвенирование ДНК может использоваться для определения последовательности отдельных гены, более крупные генетические области (т.е. кластеры генов или опероны), полные хромосомы или целые геномы любого организма. Секвенирование ДНК также является наиболее эффективным способом косвенного секвенирования РНК или же белки (через их открытые рамки для чтения). Фактически, секвенирование ДНК стало ключевой технологией во многих областях биологии и других наук, таких как медицина, криминалистика, и антропология.
Молекулярная биология
Секвенирование используется в молекулярная биология изучать геномы и белки, которые они кодируют. Информация, полученная с помощью секвенирования, позволяет исследователям идентифицировать изменения в генах, ассоциации с заболеваниями и фенотипами, а также определять потенциальные мишени для лекарств.
Эволюционная биология
Поскольку ДНК является информативной макромолекулой с точки зрения передачи от одного поколения к другому, секвенирование ДНК используется в эволюционная биология изучить, как связаны разные организмы и как они развивались.
Метагеномика
Поле метагеномика включает идентификацию организмов, присутствующих в водоеме, сточные воды, грязь, мусор, отфильтрованные из воздуха, или образцы мазков от организмов. Знание, какие организмы присутствуют в конкретной среде, имеет решающее значение для исследований в экология, эпидемиология, микробиология, и другие поля. Секвенирование позволяет исследователям определить, какие типы микробов могут присутствовать в микробиом, Например.
Вирусология
Поскольку большинство вирусов слишком малы, чтобы их можно было увидеть в световой микроскоп, секвенирование является одним из основных инструментов вирусологии для идентификации и изучения вируса.[8] Вирусные геномы могут быть основаны на ДНК или РНК. РНК-вирусы более чувствительны ко времени для секвенирования генома, поскольку они быстрее разлагаются в клинических образцах.[9] Традиционный Секвенирование по Сэнгеру секвенирование следующего поколения используется для секвенирования вирусов в фундаментальных и клинических исследованиях, а также для диагностики возникающих вирусных инфекций, молекулярная эпидемиология вирусных патогенов и тестирование на лекарственную устойчивость. Более 2,3 миллиона уникальных вирусных последовательностей GenBank.[8] В последнее время NGS превзошел традиционный метод Сэнгера как самый популярный подход для создания вирусных геномов.[8]
Вирусное секвенирование можно использовать во время эпидемии для определения причин вспышки. Вовремя Вспышка птичьего гриппа 1997 г., вирусное секвенирование определило, что подтип гриппа возник через перегруппировка между перепел и птица. Это привело к принятию законодательства в Гонконг этим запретили совместную продажу живых перепелов и птицу на рынке. Секвенирование вирусов также можно использовать для оценки начала вирусной вспышки с помощью молекулярные часы техника.[9]
Лекарство
Медицинские техники могут секвенировать гены (или, теоретически, полные геномы) пациентов, чтобы определить, существует ли риск генетических заболеваний. Это форма генетическое тестирование, хотя некоторые генетические тесты могут не включать секвенирование ДНК. Кроме того, секвенирование ДНК может быть полезно для определения конкретных бактерий, чтобы учесть больше точное лечение антибиотиками, тем самым снижая риск создания устойчивость к противомикробным препаратам в популяциях бактерий.[10][11][12][13][14][15]
Криминалистика
Секвенирование ДНК может использоваться вместе с ДНК-профилирование методы для судебно-медицинская идентификация[16] и тестирование на отцовство. За последние несколько десятилетий тестирование ДНК претерпело огромные изменения, в конечном итоге связав отпечаток ДНК с тем, что находится под следствием. Образцы ДНК в отпечатках пальцев, слюне, волосяных фолликулах и т. Д. Уникально отделяют каждый живой организм от другого. Тестирование ДНК - это метод, который может обнаруживать определенные геномы в цепи ДНК для создания уникального и индивидуального рисунка.
Четыре канонических основы
Каноническая структура ДНК имеет четыре основания: тимин (Т), аденин (А), цитозин (C) и гуанин (ГРАММ). Секвенирование ДНК - это определение физического порядка этих оснований в молекуле ДНК. Однако есть много других оснований, которые могут присутствовать в молекуле. У некоторых вирусов (в частности, бактериофаг) цитозин может быть заменен гидроксиметилом или гидроксиметил цитозином глюкозы.[17] В ДНК млекопитающих вариантные основания с метил могут быть обнаружены группы или фосфосульфат.[18][19] В зависимости от метода секвенирования конкретная модификация, например, 5mC (5 метилцитозин) часто встречается у людей, может быть обнаружен или не обнаружен.[20]
История
Открытие структуры и функции ДНК
Дезоксирибонуклеиновая кислота (ДНК) был впервые обнаружен и изолирован Фридрих Мишер в 1869 году, но он оставался недостаточно изученным в течение многих десятилетий, потому что считалось, что белки, а не ДНК содержат генетический образец жизни. Ситуация изменилась после 1944 года в результате некоторых экспериментов Освальд Эйвери, Колин МакЛауд, и Маклин Маккарти демонстрация того, что очищенная ДНК может превращать один штамм бактерий в другой. Это был первый случай, когда ДНК была продемонстрирована способность трансформировать свойства клеток.
В 1953 г. Джеймс Уотсон и Фрэнсис Крик выдвинули свои двойная спираль модель ДНК, основанная на кристаллизованный рентгеновский снимок структуры, изучаемые Розалинд Франклин. Согласно модели, ДНК состоит из двух цепей нуклеотидов, намотанных друг на друга, связанных водородными связями и движущихся в противоположных направлениях. Каждая цепь состоит из четырех комплементарных нуклеотидов - аденина (A), цитозина (C), гуанина (G) и тимина (T) - с A на одной цепи всегда в паре с T на другой, а C всегда в паре с G. Они предположили, что такая структура позволяет использовать каждую нить для реконструкции другой, что является центральной идеей для передачи наследственной информации между поколениями.[21]

Основы секвенирования белков были впервые заложены работами Фредерик Сэнгер который к 1955 г. завершил последовательность всех аминокислот в инсулин, небольшой белок, секретируемый поджелудочной железой. Это стало первым убедительным доказательством того, что белки были химическими образованиями с определенным молекулярным паттерном, а не случайной смесью веществ, взвешенных в жидкости. Успех Сэнгера в секвенировании инсулина вдохновил исследователей-рентгеновских кристаллографов, включая Уотсона и Крика, которые к настоящему времени пытались понять, как ДНК управляет образованием белков внутри клетки. Вскоре после посещения серии лекций, прочитанных Фредериком Сэнгером в октябре 1954 года, Крик начал развивать теорию, согласно которой расположение нуклеотидов в ДНК определяет последовательность аминокислот в белках, что, в свою очередь, помогает определить функцию белка. Он опубликовал эту теорию в 1958 году.[22]
Секвенирование РНК
Секвенирование РНК была одной из самых ранних форм секвенирования нуклеотидов. Основным ориентиром секвенирования РНК является последовательность первого полного гена и полного генома Бактериофаг MS2, идентифицировано и опубликовано Уолтер Фирс и его коллеги в Гентский университет (Гент, Бельгия), в 1972 г.[23] и 1976.[24] Традиционные методы секвенирования РНК требуют создания кДНК молекула, которую необходимо секвенировать.[25]
Методы раннего секвенирования ДНК
Первый метод определения последовательностей ДНК включал в себя стратегию локализации специфичного удлинения праймера, установленную Рэй Ву в Корнелл Университет в 1970 г.[26] Катализ ДНК-полимеразой и специфическое мечение нуклеотидов, оба из которых занимают видное место в современных схемах секвенирования, были использованы для секвенирования когезионных концов ДНК фага лямбда.[27][28][29] Между 1970 и 1973 годами Ву, Р. Падманабхан и его коллеги продемонстрировали, что этот метод может быть использован для определения любой последовательности ДНК с использованием синтетических локально-специфичных праймеров.[30][31][32] Фредерик Сэнгер затем применили эту стратегию удлинения праймера для разработки более быстрых методов секвенирования ДНК в MRC Center, Кембридж, UK и опубликовали метод «секвенирования ДНК с помощью ингибиторов обрыва цепи» в 1977 году.[33] Уолтер Гилберт и Аллан Максам в Гарвард также разработаны методы секвенирования, в том числе один для «секвенирования ДНК путем химической деградации».[34][35] В 1973 году Гилберт и Максам сообщили о последовательности из 24 пар оснований, используя метод, известный как анализ блуждающих точек.[36] Достижениям в области секвенирования способствовало одновременное развитие рекомбинантная ДНК технология, позволяющая изолировать образцы ДНК от источников, отличных от вирусов.
Секвенирование полных геномов

Первым секвенированным полным ДНК геномом был геном бактериофаг φX174 в 1977 г.[37] Совет медицинских исследований ученые расшифровали полную последовательность ДНК Вирус Эпштейна-Барра в 1984 году обнаружено, что он содержит 172 282 нуклеотида. Завершение последовательности ознаменовало собой важный поворотный момент в секвенировании ДНК, поскольку это было достигнуто без предварительного знания генетического профиля вируса.[38]
Нерадиоактивный метод переноса молекул ДНК из реакционных смесей секвенирования на иммобилизирующую матрицу во время электрофореза был разработан Полом с сотрудниками в начале 1980-х годов.[39][40] Затем последовала коммерциализация секвенатора ДНК "Система прямого блоттинга-электрофореза GATC 1500" компанией GATC Biotech, которая интенсивно использовалась в рамках программы секвенирования генома ЕС, полная последовательность ДНК дрожжей Saccharomyces cerevisiae хромосома II.[41] Лерой Э. Худлаборатория в Калифорнийский технологический институт анонсировала первую полуавтоматическую машину для секвенирования ДНК в 1986 году.[42] Затем последовали Прикладные биосистемы'маркетинг первой полностью автоматизированной машины для секвенирования, ABI 370, в 1987 году и компанией Dupont's Genesis 2000[43] в котором использован новый метод флуоресцентного мечения, позволяющий идентифицировать все четыре дидезоксинуклеотида на одной дорожке. К 1990 году США Национальные институты здоровья (NIH) начали крупномасштабные испытания секвенирования Mycoplasma capricolum, кишечная палочка, Caenorhabditis elegans, и Saccharomyces cerevisiae по цене 0,75 доллара США за базу. Между тем, секвенирование человеческого кДНК последовательности, называемые выраженные теги последовательности началось в Крейг Вентерлаборатории, попытка уловить кодовую долю человеческий геном.[44] В 1995 году Вентер, Гамильтон Смити коллеги из Институт геномных исследований (TIGR) опубликовал первый полный геном свободноживущего организма, бактерии Haemophilus influenzae. Круговая хромосома содержит 1830137 оснований и ее публикация в журнале Science[45] ознаменовало первое опубликованное использование полногеномного секвенирования с использованием дробовика, устраняющее необходимость в первоначальных усилиях по картированию.
К 2001 году методы секвенирования с дробовиком были использованы для получения черновой последовательности генома человека.[46][47]
Методы высокопроизводительного секвенирования (HTS)
В середине-конце 1990-х годов было разработано несколько новых методов секвенирования ДНК, которые были реализованы в коммерческих целях. Секвенаторы ДНК к 2000 году. Вместе они назывались методами секвенирования «следующего поколения» или «второго поколения» (NGS), чтобы отличать их от более ранних методов, включая Секвенирование по Сэнгеру. В отличие от первого поколения секвенирования, технология NGS обычно характеризуется высокой масштабируемостью, что позволяет секвенировать весь геном сразу. Обычно это достигается путем фрагментации генома на мелкие кусочки, случайной выборки фрагмента и его секвенирования с использованием одной из множества технологий, таких как описанные ниже. Целый геном возможен, потому что несколько фрагментов секвенируются одновременно (давая название «массово-параллельное» секвенирование) в автоматизированном процессе.
Технология NGS предоставила исследователям огромные возможности для поиска информации о здоровье, антропологам - изучить происхождение человека и стала катализатором "Персонализированная медицина"движение. Однако это также открыло дверь к большему количеству ошибок. Существует множество программных инструментов для проведения вычислительного анализа данных NGS, каждый со своим собственным алгоритмом. Даже параметры в одном программном пакете могут изменить результат анализ. Кроме того, большой объем данных, полученных с помощью секвенирования ДНК, также потребовал разработки новых методов и программ для анализа последовательностей. Для решения этих проблем были предприняты несколько попыток разработки стандартов в области NGS, большинство из которых были небольшие усилия, предпринимаемые отдельными лабораториями. Совсем недавно крупная организованная работа, финансируемая FDA, завершилась Биокомпьютер стандарт.
26 октября 1990 г. Роджер Цзянь, Pepi Ross, Margaret Fahnestock и Allan J Johnston подали патент, описывающий пошаговое («основание за основанием») секвенирование со съемными 3'-блокаторами на массивах ДНК (блоты и отдельные молекулы ДНК).[48]В 1996 г. Пол Нюрен и его ученик Мостафа Ронаги в Королевском технологическом институте в Стокгольм опубликовали свой метод пиросеквенирование.[49]
1 апреля 1997 г. Паскаль Майер и Лоран Фаринелли подали во Всемирную организацию интеллектуальной собственности патенты, описывающие секвенирование колоний ДНК.[50] Подготовка образца ДНК и случайная поверхность-полимеразной цепной реакции (ПЦР) методы компоновки, описанные в этом патенте, в сочетании с методом «основание за основанием» Роджера Цзена и др., Теперь реализованы в ИллюминаСеквенсоры генома Hi-Seq.
В 1998 году Фил Грин и Брент Юинг из Вашингтонского университета описали свои показатель качества phred для анализа данных секвенсора,[51] метод анализа, который получил широкое распространение и до сих пор остается наиболее распространенным показателем для оценки точности платформы секвенирования.[52]
Lynx Therapeutics опубликована и продана массовая параллельная последовательность подписей (MPSS) в 2000 году. Этот метод включал параллельную, опосредованную адаптером / лигированием технологию секвенирования на основе гранул и служил первым коммерчески доступным методом секвенирования «следующего поколения», хотя Секвенаторы ДНК были проданы независимым лабораториям.[53]
Основные методы
Секвенирование Максама-Гилберта
Аллан Максам и Уолтер Гилберт опубликовал метод секвенирования ДНК в 1977 году, основанный на химической модификации ДНК и последующем расщеплении по определенным основаниям.[34] Этот метод, также известный как химическое секвенирование, позволял использовать очищенные образцы двухцепочечной ДНК без дальнейшего клонирования. Использование радиоактивного мечения в этом методе и его техническая сложность препятствовали его широкому использованию после того, как методы Сэнгера были усовершенствованы.
Секвенирование Maxam-Gilbert требует радиоактивного мечения на одном 5'-конце ДНК и очистки фрагмента ДНК, который нужно секвенировать. Затем химическая обработка приводит к разрыву небольшого количества одного или двух из четырех нуклеотидных оснований в каждой из четырех реакций (G, A + G, C, C + T). Концентрация модифицирующих химикатов контролируется для введения в среднем одной модификации на молекулу ДНК. Таким образом, образуется серия меченых фрагментов от конца с радиоактивной меткой до первого «разрезанного» сайта в каждой молекуле. Фрагменты в четырех реакциях подвергают электрофорезу бок о бок в денатурирующих акриламидных гелях для разделения по размеру. Для визуализации фрагментов гель экспонируют на рентгеновской пленке для авторадиографии, получая серию темных полос, каждая из которых соответствует радиоактивно меченному фрагменту ДНК, из которых можно сделать вывод о последовательности.[34]
Методы завершения цепи
В метод обрыва цепи разработан Фредерик Сэнгер и сотрудники в 1977 году вскоре стали предпочтительным методом из-за его относительной простоты и надежности.[33][54] При изобретении метода обрыва цепи использовалось меньше токсичных химикатов и меньшее количество радиоактивности, чем в методе Максама и Гилберта. Из-за своей сравнительной простоты метод Сэнгера вскоре был автоматизирован и стал методом, использованным в первом поколении Секвенаторы ДНК.
Секвенирование по Сэнгеру - метод, который преобладал с 1980-х до середины 2000-х годов. За этот период были достигнуты большие успехи в таких технологиях, как флуоресцентное маркирование, капиллярный электрофорез и общая автоматизация. Эти разработки позволили значительно повысить эффективность секвенирования, что привело к снижению затрат. Метод Зангера в форме массового производства - это технология, которая позволила получить первый геном человека в 2001 году, вступив в эпоху геномика. Однако в конце десятилетия на рынке появились радикально иные подходы, в результате чего стоимость генома снизилась со 100 миллионов долларов в 2001 году до 10 тысяч долларов в 2011 году.[55]
Крупномасштабное секвенирование и de novo последовательность действий
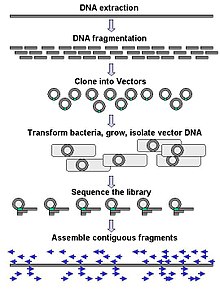
Крупномасштабное секвенирование часто направлено на секвенирование очень длинных участков ДНК, например целых хромосомы, хотя крупномасштабное секвенирование также можно использовать для генерации очень большого количества коротких последовательностей, таких как фаговый дисплей. Для более длинных целей, таких как хромосомы, общие подходы состоят в разрезании (с рестрикционные ферменты) или разрезание (с помощью механических сил) больших фрагментов ДНК на более короткие фрагменты ДНК. Затем фрагментированная ДНК может быть клонированный в Вектор ДНК и амплифицирован в бактериальном хозяине, таком как кишечная палочка. Короткие фрагменты ДНК, очищенные из отдельных бактериальных колоний, индивидуально секвенируются и собран в электронном виде в одну длинную непрерывную последовательность. Исследования показали, что добавление этапа выбора размера для сбора фрагментов ДНК одинакового размера может повысить эффективность секвенирования и точность сборки генома. В этих исследованиях автоматическая калибровка оказалась более воспроизводимой и точной, чем ручная калибровка геля.[56][57][58]
Период, термин "de novo «секвенирование» конкретно относится к методам, используемым для определения последовательности ДНК без ранее известной последовательности. De novo переводится с латыни как «с самого начала». Пробелы в собранной последовательности могут быть заполнены грунтовка ходьба. Различные стратегии имеют разные компромиссы в скорости и точности; методы дробовика часто используются для секвенирования больших геномов, но их сборка сложна и трудна, особенно с последовательность повторений часто вызывает пробелы в сборке генома.
Большинство подходов к секвенированию используют in vitro этап клонирования для амплификации отдельных молекул ДНК, потому что их методы молекулярного обнаружения недостаточно чувствительны для секвенирования отдельных молекул. ПЦР эмульсии[59] изолирует отдельные молекулы ДНК вместе с покрытыми праймером шариками в водных каплях в масляной фазе. А полимеразной цепной реакции (ПЦР) затем покрывает каждую гранулу клональными копиями молекулы ДНК с последующей иммобилизацией для последующего секвенирования. ПЦР эмульсии используется в методах, разработанных Marguilis et al. (коммерциализировано 454 Науки о жизни), Шендуре и Поррека и др. (также известный как "секвенирование полонии") и SOLiD секвенирование, (разработан Agencourt, потом Прикладные биосистемы, сейчас же Технологии жизни).[60][61][62] Эмульсионная ПЦР также используется в платформах GemCode и Chromium, разработанных 10x Геномика.[63]
Секвенирование дробовика
Секвенирование дробовиком - это метод секвенирования, разработанный для анализа последовательностей ДНК длиной более 1000 пар оснований, вплоть до целых хромосом. Этот метод требует, чтобы целевая ДНК была разбита на случайные фрагменты. После секвенирования отдельных фрагментов последовательности могут быть повторно собраны на основе их перекрывающихся областей.[64]
Методы с высокой пропускной способностью
Высокопроизводительное секвенирование, которое включает методы секвенирования следующего поколения «короткое чтение» и «длинное считывание» третьего поколения,[nt 1] применяется к секвенированию экзома, секвенированию генома, повторному секвенированию генома, транскриптом профилирование (РНК-Seq), ДНК-белковые взаимодействия (ChIP-секвенирование), и эпигеном характеристика.[65] Повторное секвенирование необходимо, потому что геном одного человека вида не будет указывать на все вариации генома среди других особей того же вида.
Высокий спрос на низкозатратное секвенирование привело к развитию технологий высокопроизводительного секвенирования, которые распараллеливать процесс секвенирования, производящий одновременно тысячи или миллионы последовательностей.[66][67][68] Технологии высокопроизводительного секвенирования призваны снизить стоимость секвенирования ДНК по сравнению с тем, что возможно при использовании стандартных методов определения терминатора с использованием красителя.[69] При секвенировании со сверхвысокой пропускной способностью параллельно может выполняться до 500 000 операций секвенирования путем синтеза.[70][71][72] Такие технологии позволили секвенировать весь геном человека всего за один день.[73] По состоянию на 2019 год[Обновить], корпоративные лидеры в разработке продуктов для высокопроизводительного секвенирования, включая Иллюмина, Qiagen и ThermoFisher Scientific.[73]
| Метод | Прочитать длину | Точность (однократное чтение, а не консенсус) | Чтений за прогон | Время на запуск | Стоимость на 1 миллиард баз (в долларах США) | Преимущества | Недостатки |
|---|---|---|---|---|---|---|---|
| Секвенирование одной молекулы в реальном времени (Pacific Biosciences) | 30 000 б.п. (N50); | 87% точность считывания[79] | 4 000 000 на ячейку SMRT Sequel 2, 100–200 гигабаз[76][80][81] | От 30 минут до 20 часов[76][82] | $7.2-$43.3 | Быстрый. Обнаруживает 4 мкКл, 5 мкКл, 6 мА.[83] | Умеренная пропускная способность. Оборудование может быть очень дорогим. |
| Ионный полупроводник (секвенирование ионного торрента) | до 600 п.н.[84] | 99.6%[85] | до 80 миллионов | 2 часа | $66.8-$950 | Менее дорогое оборудование. Быстрый. | Гомополимерные ошибки. |
| Пиросеквенирование (454) | 700 п.н. | 99.9% | 1 миллион | 24 часа | $10,000 | Длинный размер чтения. Быстрый. | Прогоны дорогие. Ошибки гомополимера. |
| Секвенирование путем синтеза (Illumina) | MiniSeq, NextSeq: 75–300 п.н .; MiSeq: 50–600 п.н .; HiSeq 2500: 50–500 п.н .; HiSeq 3/4000: 50–300 пар оснований; HiSeq X: 300 п.н. | 99,9% (Phred30) | MiniSeq / MiSeq: 1–25 миллионов; NextSeq: 130-00 миллионов; HiSeq 2500: 300 миллионов - 2 миллиарда; HiSeq 3/4000 2,5 миллиарда; HiSeq X: 3 миллиарда | От 1 до 11 дней, в зависимости от секвенсора и указанной длины чтения[86] | От 5 до 150 долларов | Возможность высокого выхода секвенирования в зависимости от модели секвенатора и желаемого применения. | Оборудование может быть очень дорогим. Требуется высокая концентрация ДНК. |
| Комбинаторный синтез якоря зонда (cPAS-BGI / MGI) | BGISEQ-50: 35-50 бп; MGISEQ 200: 50-200 бп; BGISEQ-500, MGISEQ-2000: 50-300 б.п.[87] | 99,9% (Phred30) | БГИСЭК-50: 160М; MGISEQ 200: 300M; BGISEQ-500: 1300M на проточную кювету; MGISEQ-2000: проточная кювета FCS 375M, проточная кювета FCL 1500M на каждую проточную кювету. | От 1 до 9 дней в зависимости от прибора, длины считывания и количества одновременных запусков проточных кювет. | $5– $120 | ||
| Секвенирование лигированием (SOLiD-секвенирование) | 50 + 35 или 50 + 50 б.п. | 99.9% | От 1,2 до 1,4 миллиарда | 1-2 недели | $60–130 | Низкая стоимость базы. | Медленнее, чем другие методы. Имеет проблемы с последовательностью палиндромных последовательностей.[88] |
| Секвенирование нанопор | Зависит от подготовки библиотеки, а не от устройства, поэтому пользователь выбирает длину чтения (сообщается до 2272580 битов).[89]). | ~ 92–97% однократного чтения | зависит от длины чтения, выбранной пользователем | данные передаются в реальном времени. Выберите от 1 минуты до 48 часов | $7–100 | Самый длинный человек читает. Доступное сообщество пользователей. Портативный (размером с ладонь). | Более низкая пропускная способность, чем у других машин, точность однократного считывания за 90 секунд. |
| Секвенирование GenapSys | Около 150 п.н. односторонний | 99,9% (Phred30) | От 1 до 16 миллионов | Около 24 часов | $667 | Низкая стоимость инструмента (10 000 $) | |
| Обрыв цепи (секвенирование по Сэнгеру) | От 400 до 900 п.н. | 99.9% | Нет данных | От 20 минут до 3 часов | $2,400,000 | Полезно для многих приложений. | Более дорогой и непрактичный для больших проектов секвенирования. Этот метод также требует трудоемкого этапа клонирования плазмиды или ПЦР. |
Долговременные методы секвенирования
Секвенирование одиночных молекул в реальном времени (SMRT)
Секвенирование SMRT основано на методе секвенирования путем синтеза. ДНК синтезируется в волноводах нулевой моды (ZMW) - небольших хорошо похожих на сосуды контейнерах с инструментами для улавливания, расположенными на дне скважины.Секвенирование выполняется с использованием немодифицированной полимеразы (прикрепленной к основанию ZMW) и флуоресцентно меченных нуклеотидов, свободно протекающих в растворе. Лунки сконструированы таким образом, что регистрируется только флуоресценция, возникающая на дне лунки. Флуоресцентная метка отделяется от нуклеотида при его включении в цепь ДНК, оставляя немодифицированную цепь ДНК. В соответствии с Тихоокеанские биологические науки (PacBio), разработчик технологии SMRT, эта методология позволяет обнаруживать модификации нуклеотидов (например, метилирование цитозина). Это происходит благодаря наблюдению за кинетикой полимеразы. Этот подход позволяет считывать 20 000 нуклеотидов и более при средней длине считывания 5 килобаз.[80][90] В 2015 году Pacific Biosciences объявила о запуске нового инструмента для секвенирования под названием Sequel System с 1 миллионом ZMW по сравнению с 150 000 ZMW в инструменте PacBio RS II.[91][92] SMRT-секвенирование называется "третье поколение"или" долгосрочное "секвенирование.
Секвенирование ДНК нанопор
ДНК, проходящая через нанопору, изменяет свой ионный ток. Это изменение зависит от формы, размера и длины последовательности ДНК. Каждый тип нуклеотида блокирует поток ионов через пору на разный период времени. Метод не требует модифицированных нуклеотидов и выполняется в режиме реального времени. Секвенирование нанопор называется "третье поколение"или" долгое чтение "секвенирования, наряду с секвенированием SMRT.
Ранние промышленные исследования этого метода были основаны на методе, называемом «секвенирование экзонуклеаз», при котором считывание электрических сигналов происходило при прохождении нуклеотидов. альфа (α) -гемолизин поры ковалентно связаны с циклодекстрин.[93] Однако последующий коммерческий метод, «секвенирование цепи», секвенировал основания ДНК в интактной цепи.
Двумя основными направлениями развития секвенирования нанопор являются твердотельное секвенирование нанопор и секвенирование нанопор на основе белков. При секвенировании белковых нанопор используются мембранные белковые комплексы, такие как α-гемолизин, MspA (Микобактерии смегматис Porin A) или CssG, которые демонстрируют большие перспективы, учитывая их способность различать отдельные нуклеотиды и группы.[94] Напротив, в твердотельном секвенировании нанопор используются синтетические материалы, такие как нитрид кремния и оксид алюминия, и они предпочтительны из-за их превосходных механических свойств, термической и химической стабильности.[95] Метод изготовления важен для этого типа секвенирования, учитывая, что массив нанопор может содержать сотни пор с диаметром менее восьми нанометров.[94]
Эта концепция возникла из идеи, что одноцепочечные молекулы ДНК или РНК могут электрофоретически управляться в строгой линейной последовательности через биологическую пору, размер которой может составлять менее восьми нанометров, и может быть обнаружен при условии, что молекулы выделяют ионный ток при движении через поры. Пора содержит область обнаружения, способную распознавать разные основания, при этом каждая база генерирует различные временные сигналы, соответствующие последовательности оснований, когда они пересекают пору, которые затем оцениваются.[95] Точный контроль над транспортом ДНК через поры имеет решающее значение для успеха. Различные ферменты, такие как экзонуклеазы и полимеразы, были использованы для смягчения этого процесса, располагая их возле входа в поры.[96]
Краткосрочные методы секвенирования
Массивно-параллельное упорядочение сигнатур (MPSS)
Первая из высокопроизводительных технологий секвенирования, массовая параллельная последовательность подписей (или MPSS), была разработана в 1990-х годах в Lynx Therapeutics, компании, основанной в 1992 году Сидней Бреннер и Сэм Элетр. MPSS был методом на основе шариков, который использовал комплексный подход лигирования адаптера с последующим декодированием адаптера, считывая последовательность с шагом в четыре нуклеотида. Этот метод сделал его чувствительным к систематическому смещению или потере конкретных последовательностей. Поскольку технология была настолько сложной, MPSS выполнялась только «внутри компании» Lynx Therapeutics, и никакие машины для секвенирования ДНК не продавались независимым лабораториям. Lynx Therapeutics слилась с Solexa (позже была приобретена Иллюмина) в 2004 г., что привело к развитию секвенирования путем синтеза, более простого подхода, приобретенного из Прогнозирующая медицина Manteia, что сделало MPSS устаревшим. Однако основные свойства вывода MPSS были типичными для более поздних типов данных с высокой пропускной способностью, включая сотни тысяч коротких последовательностей ДНК. В случае MPSS они обычно использовались для секвенирования. кДНК для измерения экспрессия гена уровни.[53]
Секвенирование полонии
В секвенирование полонии метод, разработанный в лаборатории Джордж М. Черч в Гарварде, была одной из первых систем секвенирования с высокой пропускной способностью и использовалась для секвенирования полного Кишечная палочка геном в 2005 году.[97] Он объединил библиотеку парных тегов in vitro с эмульсионной ПЦР, автоматизированным микроскопом и химией секвенирования на основе лигирования для секвенирования Кишечная палочка генома с точностью> 99,9999% и стоимостью примерно 1/9 от секвенирования по Сэнгеру.[97] Лицензия на технологию была передана Agencourt Biosciences, впоследствии она была передана в Agencourt Personal Genomics и в конечном итоге включена в Прикладные биосистемы Платформа SOLiD. Позднее Applied Biosystems была приобретена Технологии жизни, теперь часть Thermo Fisher Scientific.
454 пиросеквенирование
Распараллеленная версия пиросеквенирование был разработан 454 Науки о жизни, который с тех пор был приобретен Рош Диагностика. Этот метод амплифицирует ДНК внутри капель воды в масляном растворе (эмульсионная ПЦР), при этом каждая капля содержит одну матрицу ДНК, прикрепленную к одной покрытой праймером бусине, которая затем образует клональную колонию. Секвенсорная машина содержит много пиколитр-объемные лунки, каждая из которых содержит одну гранулу и ферменты секвенирования. Пиросеквенирование использует люцифераза для генерации света для обнаружения отдельных нуклеотидов, добавленных к зарождающейся ДНК, и объединенные данные используются для создания последовательности читает.[60] Эта технология обеспечивает промежуточную длину чтения и цену за базу по сравнению с секвенированием по Сэнгеру с одной стороны и Solexa и SOLiD с другой.[69]
Секвенирование Illumina (Solexa)
Солекса, теперь часть Иллюмина, была основана Шанкар Баласубраманян и Дэвид Кленерман в 1998 году и разработал метод секвенирования, основанный на технологии обратимых терминаторов красителей и инженерных полимераз.[98] Концепция химии с обратимым терминированием была изобретена Бруно Канардом и Саймоном Сарфати в Институте Пастера в Париже.[99][100] Он был разработан внутри компании Solexa теми, кто указан в соответствующих патентах. В 2004 году Solexa приобрела компанию. Прогнозирующая медицина Manteia чтобы получить технологию массового параллельного секвенирования, изобретенную в 1997 году Паскалем Майером и Лораном Фаринелли.[50] Он основан на «кластерах ДНК» или «колониях ДНК», который включает клональную амплификацию ДНК на поверхности. Кластерная технология была приобретена совместно с Lynx Therapeutics of California. Позже Solexa Ltd. объединилась с Lynx и образовала Solexa Inc.
В этом методе молекулы ДНК и праймеры сначала прикрепляются к предметному стеклу или проточной ячейке и амплифицируются с помощью полимераза так что образуются локальные клональные колонии ДНК, позже названные «кластерами ДНК». Для определения последовательности добавляют четыре типа оснований обратимых терминаторов (RT-основания) и смывают невключенные нуклеотиды. Камера делает снимки флуоресцентно маркированный нуклеотиды. Затем краситель вместе с концевым 3'-блокатором химически удаляется из ДНК, позволяя начать следующий цикл. В отличие от пиросеквенирования, цепи ДНК удлиняются на один нуклеотид за раз, и получение изображения может выполняться с задержкой, что позволяет захватывать очень большие массивы колоний ДНК с помощью последовательных изображений, полученных с одной камеры.
Разделение ферментативной реакции и захвата изображения обеспечивает оптимальную производительность и теоретически неограниченную емкость секвенирования. Таким образом, при оптимальной конфигурации максимально достижимая производительность инструмента определяется исключительно коэффициентом аналого-цифрового преобразования камеры, умноженным на количество камер и разделенным на количество пикселей на колонию ДНК, необходимое для их оптимальной визуализации (приблизительно 10 пикселей / колония). В 2012 году с камерами, работающими с частотой аналого-цифрового преобразования более 10 МГц и доступной оптикой, жидкостями и ферментными средствами, пропускная способность может быть кратной 1 миллиону нуклеотидов в секунду, что примерно соответствует 1 эквиваленту генома человека при 1x покрытие в час на инструмент, и 1 геном человека повторно секвенировали (примерно с 30-кратным увеличением) в день на каждый инструмент (оборудованный единственной камерой).[101]
Комбинаторный синтез якоря зонда (cPAS)
Этот метод является усовершенствованной модификацией комбинаторной технологии лигирования якоря зонда (cPAL), описанной Полная геномика[102] которая с тех пор стала частью китайской геномной компании BGI в 2013.[103] Обе компании усовершенствовали технологию, чтобы обеспечить большую длину чтения, сокращение времени реакции и более быстрое получение результатов. Кроме того, данные теперь генерируются как непрерывные полноразмерные чтения в стандартном формате файла FASTQ и могут использоваться как есть в большинстве конвейеров биоинформатического анализа на основе короткого чтения.[104][нужна цитата]
В основе этой высокопроизводительной технологии секвенирования лежат две технологии: Наношарики ДНК (DNB) и узорчатые массивы для прикрепления наношаров к твердой поверхности.[102] ДНК-наношарики просто формируются путем денатурирования двухцепочечных библиотек с лигированием адаптеров и лигирования прямой цепи только со шпоночным олигонуклеотидом с образованием круга оцДНК. Достоверные копии кругов, содержащих вставку ДНК, производятся с использованием амплификации вращающегося круга, которая генерирует примерно 300–500 копий. Длинная цепь оцДНК складывается сама по себе, образуя трехмерную структуру наношара, которая имеет диаметр примерно 220 нм. Создание DNB заменяет необходимость создания ПЦР-копий библиотеки на проточной кювете и, как таковое, может удалить большую часть повторяющихся считываний, лигирования адаптер-адаптер и ошибок, вызванных ПЦР.[104][нужна цитата]
Узорчатый массив положительно заряженных пятен создается с помощью методов фотолитографии и травления с последующей химической модификацией для создания проточной ячейки секвенирования. Каждое пятно на проточной кювете имеет диаметр примерно 250 нм, разделено 700 нм (от центра к центру) и позволяет легко прикрепить один отрицательно заряженный DNB к проточной кювете и, таким образом, уменьшить скопление или чрезмерную кластеризацию на проточной кювете.[102][нужна цитата]
Затем выполняется секвенирование путем добавления олигонуклеотидного зонда, который присоединяется в комбинации к определенным сайтам внутри DNB. Зонд действует как якорь, который затем позволяет одному из четырех одиночных обратимо инактивированных меченых нуклеотидов связываться после прохождения через проточную кювету. Несвязанные нуклеотиды вымываются перед лазерным возбуждением прикрепленных меток, затем излучают флуоресценцию, и сигнал улавливается камерами, которые преобразуются в цифровой выход для определения основания. Присоединенное основание имеет свой терминатор и метку, химически расщепленную по завершении цикла. Цикл повторяется с другим потоком свободных меченых нуклеотидов через проточную ячейку, чтобы позволить следующему нуклеотиду связываться и улавливать его сигнал. Этот процесс выполняется несколько раз (обычно от 50 до 300 раз) для определения последовательности вставленного фрагмента ДНК со скоростью примерно 40 миллионов нуклеотидов в секунду по состоянию на 2018 год.[нужна цитата]
SOLiD секвенирование

Прикладные биосистемы'(теперь Технологии жизни бренд) SOLiD Technology использует секвенирование лигированием. Здесь пул всех возможных олигонуклеотидов фиксированной длины помечен в соответствии с положением в последовательности. Олигонуклеотиды отжигаются и лигируются; предпочтительное лигирование ДНК-лигаза для сопоставления последовательностей приводит к сигналу, информативному о нуклеотиде в этом положении. Каждая база в шаблоне дважды упорядочивается, и полученные данные декодируются в соответствии с 2 базовая кодировка Схема, используемая в этом методе. Перед секвенированием ДНК амплифицируют с помощью эмульсионной ПЦР. Полученные шарики, каждая из которых содержит отдельные копии одной и той же молекулы ДНК, помещают на предметное стекло.[105] В результате получаются последовательности количества и длины, сопоставимые с секвенированием Illumina.[69] Этот секвенирование лигированием Сообщалось, что метод имеет некоторые проблемы с секвенированием палиндромных последовательностей.[88]
Секвенирование полупроводников Ion Torrent
Ion Torrent Systems Inc. (в настоящее время принадлежит Технологии жизни) разработал систему, основанную на использовании стандартной химии секвенирования, но с новой системой обнаружения на основе полупроводников. Этот метод секвенирования основан на обнаружении ионы водорода которые выпущены во время полимеризация из ДНК, в отличие от оптических методов, используемых в других системах секвенирования. Микролунка, содержащая матричную цепь ДНК, подлежащую секвенированию, заполняется одним типом нуклеотид. Если введенный нуклеотид дополнительный к ведущему нуклеотиду матрицы он включается в растущую комплементарную цепь. Это вызывает высвобождение иона водорода, который запускает сверхчувствительный ионный датчик, что указывает на то, что произошла реакция. Если гомополимер в матричной последовательности присутствуют повторы, несколько нуклеотидов будут включены в один цикл. Это приводит к соответствующему количеству высвобождаемых водородов и пропорционально более высокому электронному сигналу.[106]
Секвенирование наношаров ДНК
Секвенирование наношаров ДНК это тип высокопроизводительной технологии секвенирования, используемой для определения всего геномная последовательность организма. Компания Полная геномика использует эту технологию для секвенирования образцов, представленных независимыми исследователями. Метод использует репликация катящегося круга для амплификации небольших фрагментов геномной ДНК в наношарики ДНК. Затем для определения нуклеотидной последовательности используется свободное секвенирование путем лигирования.[107] Этот метод секвенирования ДНК позволяет секвенировать большое количество наношаров ДНК за цикл и при низкой реагент затраты по сравнению с другими платформами секвенирования с высокой пропускной способностью.[108] Однако только короткие последовательности ДНК определяются из каждого наношара ДНК, что позволяет отображать короткие чтения на эталонный геном трудно.[107] Эта технология использовалась для нескольких проектов по секвенированию генома и планируется использовать в других целях.[109]
Секвенирование одиночных молекул с помощью Heliscope
Хелископическое секвенирование - метод секвенирования одной молекулы, разработанный Helicos Biosciences. Он использует фрагменты ДНК с добавленными адаптерами хвоста поли-А, которые прикреплены к поверхности проточной ячейки. Следующие шаги включают основанное на расширении секвенирование с циклической промывкой проточной кюветы флуоресцентно меченными нуклеотидами (по одному типу нуклеотидов за раз, как в методе Сенгера). Считывание выполняется секвенсором Heliscope.[110][111] Читки короткие, в среднем 35 б.п.[112] Что сделало эту технологию особенно новой, так это то, что она была первой в своем классе, которая секвенировала неамплифицированную ДНК, тем самым предотвращая любые ошибки чтения, связанные с этапами амплификации.[113] В 2009 году геном человека был секвенирован с помощью Heliscope, однако в 2012 году компания обанкротилась.[114]
Микрожидкостные системы
Есть две основные микрофлюидные системы, которые используются для секвенирования ДНК; капельная микрофлюидика и цифровая микрофлюидика. Микрожидкостные устройства решают многие из текущих ограничений современных систем секвенирования.
Abate et al. изучал использование микрофлюидных устройств на основе капель для секвенирования ДНК.[4] Эти устройства способны формировать и обрабатывать капли размером с пиколитр со скоростью тысячи в секунду. Устройства созданы из полидиметилсилоксан (ПДМС)и использовал резонансный перенос энергии Форстера, FRET анализы для чтения последовательностей ДНК, заключенных в капельках. Каждая позиция в массиве проверена на конкретную 15 базовую последовательность.[4]
Fair et al. использовали цифровые микрофлюидные устройства для изучения ДНК пиросеквенирование.[115] Существенные преимущества включают портативность устройства, объем реагента, скорость анализа, возможности массового производства и высокую производительность. Это исследование предоставило доказательство концепции, показывающей, что цифровые устройства можно использовать для пиросеквенирования; исследование включало использование синтеза, который включает расширение ферментов и добавление меченых нуклеотидов.[115]
Boles et al. также изучал пиросеквенирование на цифровых микрофлюидных устройствах.[116] Они использовали электросмачивающее устройство для создания, смешивания и разделения капель. При секвенировании используется протокол с тремя ферментами и шаблоны ДНК, закрепленные магнитными шариками. Устройство было протестировано с использованием двух протоколов и показало 100% точность на основе необработанных уровней пирограммы. Преимущества этих цифровых микрофлюидных устройств включают размер, стоимость и достижимые уровни функциональной интеграции.[116]
Исследования секвенирования ДНК с использованием микрофлюидики также могут быть применены к секвенирование РНК, используя аналогичные методы капельной микрофлюидии, такие как метод inDrops.[117] Это показывает, что многие из этих методов секвенирования ДНК можно будет применять и дальше, чтобы лучше понять геномы и транскриптомы.
Методы в разработке
Методы секвенирования ДНК, которые в настоящее время разрабатываются, включают считывание последовательности по мере прохождения цепи ДНК через нанопоры (метод, который сейчас является коммерческим, но последующие поколения, такие как твердотельные нанопоры, все еще находятся в разработке),[118][119] и методы на основе микроскопии, такие как атомно-силовая микроскопия или же просвечивающая электронная микроскопия которые используются для идентификации положений отдельных нуклеотидов в длинных фрагментах ДНК (> 5000 п.н.) путем маркировки нуклеотидов более тяжелыми элементами (например, галогенами) для визуального обнаружения и регистрации.[120][121]Технологии третьего поколения стремятся увеличить производительность и сократить время получения результата и стоимость за счет устранения необходимости в избыточных реагентах и использования процессивности ДНК-полимеразы.[122]
Туннельные токи Секвенирование ДНК
Другой подход использует измерения электрических туннельных токов через однонитевую ДНК, когда она движется через канал. В зависимости от своей электронной структуры каждая база по-разному влияет на туннельный ток,[123] позволяя различать разные базы.[124]
Использование туннельных токов может упорядочить на порядки быстрее, чем методы ионного тока, и уже достигнуто секвенирование нескольких олигомеров ДНК и микро-РНК.[125]
Секвенирование путем гибридизации
Секвенирование путем гибридизации это неферментативный метод, который использует Микрочип ДНК. Единый пул ДНК, последовательность которого необходимо определить, флуоресцентно метят и гибридизуют с массивом, содержащим известные последовательности. Сильные сигналы гибридизации от данного места на массиве идентифицируют его последовательность в секвенируемой ДНК.[126]
Этот метод секвенирования использует характеристики связывания библиотеки коротких одноцепочечных молекул ДНК (олигонуклеотидов), также называемых ДНК-зондами, для восстановления целевой последовательности ДНК. Неспецифические гибриды удаляют промыванием и элюируют ДНК-мишень.[127] Гибриды перестраиваются таким образом, чтобы можно было реконструировать последовательность ДНК. Преимущество этого типа секвенирования заключается в его способности захватывать большое количество целей с однородным покрытием.[128] Обычно требуется большое количество химикатов и стартовая ДНК. Однако с появлением гибридизации на основе растворов требуется гораздо меньше оборудования и химикатов.[127]
Секвенирование с масс-спектрометрией
Масс-спектрометрии может использоваться для определения последовательностей ДНК. Матричная лазерная десорбционно-ионизационная времяпролетная масс-спектрометрия, или MALDI-TOF MS, был специально исследован как альтернативный метод гель-электрофорезу для визуализации фрагментов ДНК. С помощью этого метода фрагменты ДНК, полученные в результате реакций секвенирования обрыва цепи, сравниваются по массе, а не по размеру. Масса каждого нуклеотида отличается от массы других, и эту разницу можно обнаружить с помощью масс-спектрометрии. Однонуклеотидные мутации во фрагменте легче обнаружить с помощью МС, чем с помощью только гель-электрофореза. MALDI-TOF MS может более легко обнаруживать различия между фрагментами РНК, поэтому исследователи могут косвенно секвенировать ДНК с помощью методов на основе MS, сначала преобразовав ее в РНК.[129]
Более высокое разрешение фрагментов ДНК, разрешенное методами на основе МС, представляет особый интерес для исследователей судебной медицины, поскольку они, возможно, захотят найти однонуклеотидные полиморфизмы в образцах ДНК человека для идентификации людей. Эти образцы могут быть сильно деградированы, поэтому судебно-медицинские эксперты часто предпочитают митохондриальная ДНК за его более высокую стабильность и приложения для изучения происхождения. Методы секвенирования на основе МС были использованы для сравнения последовательностей митохондриальной ДНК человека из образцов в Федеральное Бюро Расследований база данных[130] и из костей, найденных в братских могилах солдат Первой мировой войны.[131]
Методы раннего обрыва цепи и TOF MS продемонстрировали длину считывания до 100 пар оснований.[132] Исследователи не смогли превысить этот средний размер чтения; как и только секвенирование обрыва цепи, секвенирование ДНК на основе МС может не подходить для больших de novo секвенирование проектов. Несмотря на это, недавнее исследование действительно использовало считывание коротких последовательностей и масс-спектроскопию для сравнения однонуклеотидных полиморфизмов в патогенных Стрептококк штаммы.[133]
Микрожидкостное секвенирование по Сэнгеру
В микрофлюидных Секвенирование по Сэнгеру вся амплификация фрагментов ДНК с помощью термоциклирования, а также их разделение с помощью электрофореза выполняется на одной стеклянной пластине (примерно 10 см в диаметре), что снижает использование реагентов, а также стоимость.[134] В некоторых случаях исследователи показали, что они могут увеличить производительность обычного секвенирования с помощью микрочипов.[135] По-прежнему необходимо будет провести исследования, чтобы сделать такое использование технологий эффективным.
Методы на основе микроскопии
Этот подход позволяет непосредственно визуализировать последовательность молекул ДНК с помощью электронной микроскопии. Первая идентификация пар оснований ДНК в интактных молекулах ДНК путем ферментативного включения модифицированных оснований, которые содержат атомы с увеличенным атомным номером, прямой визуализации и идентификации индивидуально помеченных оснований в синтетической молекуле ДНК с 3272 парами оснований и вирусном геноме с 7249 парами оснований был продемонстрирован.[136]
Секвенирование RNAP
Этот метод основан на использовании РНК-полимераза (RNAP), который прикреплен к полистирол шарик. Один конец ДНК, подлежащий секвенированию, прикрепляют к другой бусине, причем обе бусинки помещают в оптические ловушки. Движение RNAP во время транскрипции сближает шарики, и их относительное расстояние изменяется, что затем может быть записано с разрешением в один нуклеотид. Последовательность выводится на основе четырех считываний с пониженными концентрациями каждого из четырех типов нуклеотидов, аналогично методу Сэнгера.[137] Сравнение проводится между областями, и информация о последовательности выводится путем сравнения областей известной последовательности с областями неизвестной последовательности.[138]
В пробирке высокопроизводительное секвенирование вирусов
Разработан метод анализа полных наборов белковые взаимодействия с использованием комбинации 454 пиросеквенирования и in vitro вирус отображение мРНК метод. В частности, этот метод ковалентно связывает интересующие белки с кодирующими их мРНК, а затем обнаруживает фрагменты мРНК с помощью обратной транскрипции. ПЦР. Затем мРНК можно амплифицировать и секвенировать. Комбинированный метод получил название IVV-HiTSeq и может выполняться в бесклеточных условиях, хотя его результаты могут не отражать in vivo условия.[139]
Базовые приготовления
Успех любого протокола секвенирования ДНК зависит от экстракции и подготовки образца ДНК или РНК из интересующего биологического материала.
- В результате успешной экстракции ДНК будет получен образец ДНК с длинными неразрушенными цепями.
- Успешная экстракция РНК даст образец РНК, который необходимо преобразовать в комплементарную ДНК (кДНК) с использованием обратной транскриптазы - ДНК-полимеразы, которая синтезирует комплементарную ДНК на основе существующих цепей РНК способом, подобным ПЦР.[140] Комплементарную ДНК затем можно обрабатывать так же, как и геномную ДНК.
В соответствии с используемой технологией секвенирования образцы, полученные в результате экстракции ДНК или РНК, требуют дополнительной подготовки. Для секвенирования по Сэнгеру перед секвенированием требуются либо процедуры клонирования, либо ПЦР. В случае методов секвенирования следующего поколения перед обработкой требуется подготовка библиотеки.[141] Оценка качества и количества нуклеиновых кислот как после экстракции, так и после подготовки библиотеки выявляет деградированные, фрагментированные и низкочистые образцы и дает высококачественные данные секвенирования.[142]
Высокопроизводительный характер современных технологий секвенирования ДНК / РНК создал проблему для масштабирования метода подготовки проб. Несколько инструментов для работы с жидкостями используются для подготовки большего количества проб с меньшим общим временем работы:
| Компания | Погрузчики жидкости / Автоматизация | Landing_url |
|---|---|---|
| Опентроны | ОпенТронс ОТ-2 | https://www.opentrons.com/ |
| Agilent | Agilent Bravo NGS | https://www.agilent.com/en/products/automated-liquid-handling/automated-liquid-handling-applications/bravo-ngs |
| Бекман Коултер | Beckman Coulter Biomek iSeries | https://www.beckman.com/liquid-handlers/biomek-i7/features |
| Eppendorf | Eppendorf epMotion 5075t | https://www.eppendorf.com/epmotion/ |
| Гамильтон | NGS STAR | http://www.hamiltonrobotics.com/ |
| ПеркинЭлмер | Sciclone G3 NGS и рабочая станция NGSx | https://www.perkinelmer.com/uk/product/sciclone-g3-ngs-workstation-cls145321 |
| Tecan | Tecan Freedom EVO NGS | https://lifesciences.tecan.com/ngs-sample-preparation |
| Hudson Robotics | Hudson Robotics СОЛО | https://hudsonrobotics.com/products/applications/automated-solutions-next-generation-sequencing-ngs/ |
Инициативы развития

В октябре 2006 г. X Prize Foundation выступил с инициативой по содействию развитию полное секвенирование генома технологии, называемые Приз Archon X, намереваясь наградить 10 миллионов долларов «первой команде, которая сможет создать устройство и использовать его для секвенирования 100 геномов человека в течение 10 дней или меньше, с точностью не более одной ошибки на каждые 100 000 секвенированных оснований, с последовательностями, точно покрывающими не менее 98% генома и при повторяющейся стоимости не более 10 000 долларов США за геном ".[143]
Каждый год Национальный институт исследования генома человека, или NHGRI, продвигает гранты на новые исследования и разработки в геномика. Гранты 2010 г. и кандидаты 2011 г. включают продолжение работы в области микрожидкостных, полониевых и методологий секвенирования на основе тяжелых оснований.[144]
Вычислительные проблемы
Описанные здесь технологии секвенирования производят необработанные данные, которые необходимо собрать в более длинные последовательности, такие как полные геномы (сборка последовательности). Для достижения этой цели существует множество вычислительных задач, таких как оценка необработанных данных последовательности, которая выполняется программами и алгоритмами, такими как Фред и Phrap. Другие проблемы, с которыми придется столкнуться повторяющийся последовательности, которые часто препятствуют полной сборке генома, поскольку они встречаются во многих местах генома. Как следствие, многие последовательности не могут быть отнесены к конкретным хромосомы. Производство необработанных данных последовательности - это только начало ее подробного биоинформатический анализ.[145] Однако были разработаны новые методы секвенирования и исправления ошибок секвенирования.[146]
Читать обрезку
Иногда необработанные считывания, производимые секвенсором, являются правильными и точными только в части их длины. Использование всего чтения может привести к появлению артефактов в последующих анализах, таких как сборка генома, вызов snp или оценка экспрессии генов. Были представлены два класса программ обрезки, основанные на классах алгоритмов, основанных на окнах или на классах алгоритмов с промежуточной суммой.[147] Это неполный список алгоритмов обрезки, доступных в настоящее время, с указанием класса алгоритмов, к которому они принадлежат:
| Название алгоритма | Тип алгоритма | Связь |
|---|---|---|
| Cutadapt[148] | Текущая сумма | Cutadapt |
| ConDeTri[149] | На основе окна | ConDeTri |
| ERNE-ФИЛЬТР[150] | Текущая сумма | ERNE-ФИЛЬТР |
| Качественный триммер FASTX | На основе окна | Качественный триммер FASTX |
| PRINSEQ[151] | На основе окна | PRINSEQ |
| Trimmomatic[152] | На основе окна | Trimmomatic |
| SolexaQA[153] | На основе окна | SolexaQA |
| SolexaQA-BWA | Текущая сумма | SolexaQA-BWA |
| Серп | На основе окна | Серп |
Этические вопросы
Эта секция нуждается в расширении. Вы можете помочь добавляя к этому. (Май 2015 г.) |
Генетика человека была включена в сферу биоэтика с начала 1970-х[154] а рост использования секвенирования ДНК (особенно высокопроизводительного секвенирования) привел к возникновению ряда этических проблем. Одна из ключевых проблем - это право собственности на ДНК человека и данные, полученные при секвенировании этой ДНК.[155] Что касается самой молекулы ДНК, ведущего судебного дела по этой теме, Мур против Регентов Калифорнийского университета (1990) постановили, что люди не имеют права собственности на выброшенные ячейки или какую-либо прибыль, полученную с использованием этих ячеек (например, в качестве запатентованных клеточная линия). Однако люди имеют право на информированное согласие на удаление и использование клеток. Что касается данных, полученных с помощью секвенирования ДНК, Мур не дает человеку никаких прав на информацию, полученную из его ДНК.[155]
Поскольку секвенирование ДНК становится все более распространенным, хранение, безопасность и совместное использование геномных данных также становятся все более важными.[155][156] Например, одна проблема заключается в том, что страховщики могут использовать геномные данные человека для изменения своей квоты в зависимости от предполагаемого будущего здоровья человека на основе его ДНК.[156][157] В мае 2008 г. Закон о недискриминации в отношении генетической информации (GINA) был подписан в США, запрещая дискриминацию на основе генетической информации в отношении медицинского страхования и занятости.[158][159] В 2012 году США Президентская комиссия по изучению биоэтических вопросов сообщили, что существующее законодательство о конфиденциальности для данных секвенирования ДНК, таких как GINA и Медицинское страхование Портативность и Акт об ответственности были недостаточны, учитывая, что данные полногеномного секвенирования были особенно чувствительными, поскольку их можно было использовать для идентификации не только человека, от которого были созданы данные, но и их родственников.[160][161]
Этические проблемы также были подняты в связи с более широким использованием скрининга генетических вариаций как у новорожденных, так и у взрослых такими компаниями, как 23andMe.[162][163] Утверждалось, что скрининг на генетические вариации может быть вредным, увеличивая беспокойство у лиц, у которых обнаружен повышенный риск заболевания.[164] Например, в одном случае, отмеченном в Время, врачи, обследующие больного ребенка на генетические варианты, решили не сообщать родителям о неродственном варианте, связанном с слабоумие из-за вреда, который это может причинить родителям.[165] Однако исследование 2011 г. Медицинский журнал Новой Англии показал, что люди, подвергшиеся профилированию риска заболевания, не проявляют повышенного уровня тревожности.[164]
Смотрите также
Примечания
- ^ «Следующее поколение» по-прежнему широко используется по состоянию на 2019 год. Например, Стрэйтон Дж., Фри Т, Сойер А., Мартин Дж. (Февраль 2019 г.). «От секвенирования по Сэнгеру к базам данных генома и не только». Биотехнологии. 66 (2): 60–63. Дои:10.2144 / btn-2019-0011. PMID 30744413.
Технологии секвенирования нового поколения (NGS) произвели революцию в геномных исследованиях. (вступительное предложение статьи)
Рекомендации
- ^ «Представляем« темную ДНК »- феномен, который может изменить наши представления об эволюции».
- ^ Бехджати С., Тарпей П.С. (декабрь 2013 г.). "Что такое секвенирование следующего поколения?". Архив болезней детства. Издание для обучения и практики. 98 (6): 236–8. Дои:10.1136 / archdischild-2013-304340. ЧВК 3841808. PMID 23986538.
- ^ Хмелецкий Дж., Мейерсон М. (14 января 2014 г.). «Секвенирование ДНК рака: что мы узнали?». Ежегодный обзор медицины. 65 (1): 63–79. Дои:10.1146 / annurev-med-060712-200152. PMID 24274178.
- ^ а б c d Abate AR, Hung T., Sperling RA, Mary P, Rotem A, Agresti JJ, et al. (Декабрь 2013). «Анализ последовательности ДНК с помощью капельной микрофлюидики». Лаборатория на чипе. 13 (24): 4864–9. Дои:10.1039 / c3lc50905b. ЧВК 4090915. PMID 24185402.
- ^ Пекин Д., Схири Ю., Барет Дж. К., Ле Корре Д., Мазутис Л., Салем С. Б. и др. (Июль 2011 г.). «Количественное и чувствительное обнаружение редких мутаций с использованием капельной микрофлюидики». Лаборатория на чипе. 11 (13): 2156–66. Дои:10.1039 / c1lc20128j. PMID 21594292.
- ^ Ольсвик О., Уолберг Дж., Петтерсон Б., Улен М., Попович Т., Ваксмут И.К., Филдс П.И. (январь 1993 г.). «Использование автоматизированного секвенирования ампликонов, генерируемых полимеразной цепной реакцией, для идентификации трех типов субъединицы В холерного токсина в штаммах Vibrio cholerae O1». J. Clin. Microbiol. 31 (1): 22–25. Дои:10.1128 / JCM.31.1.22-25.1993. ЧВК 262614. PMID 7678018.

- ^ Петтерссон Э., Лундеберг Дж., Ахмадиан А. (февраль 2009 г.). «Поколения технологий секвенирования». Геномика. 93 (2): 105–11. Дои:10.1016 / j.ygeno.2008.10.003. PMID 18992322.
- ^ а б c Кастро, Кристина; Марин, Рэйчел; Рамос, Эдвард; Нг, Терри Фей Фан (2019). «Влияние вариантной интерференции на сборку de novo для глубокого секвенирования вирусов». BMC Genomics. 21 (1): 421. bioRxiv 10.1101/815480. Дои:10.1186 / s12864-020-06801-w. ЧВК 7306937. PMID 32571214.
- ^ а б Уол, Ширли; Шаффнер, Стивен Ф .; Сабети, Пардис К. (2016). «Геномный анализ вирусных вспышек». Ежегодный обзор вирусологии. 3 (1): 173–195. Дои:10.1146 / annurev-virology-110615-035747. ЧВК 5210220. PMID 27501264.
- ^ Schleusener V, Köser CU, Beckert P, Niemann S, Feuerriegel S (2017). "Микобактерии туберкулеза прогнозирование устойчивости и классификация клонов на основе секвенирования генома: сравнение инструментов автоматического анализа ». Научный представитель. 7: 46327. Bibcode:2017НатСР ... 746327С. Дои:10.1038 / srep46327. ЧВК 7365310. PMID 28425484.
- ^ Маэ П., Эль-Азами М., Барлас П., Турно М. (2019). "Крупномасштабная оценка TBProfiler и Mykrobe для прогнозирования устойчивости к антибиотикам в Микобактерии туберкулеза". PeerJ. 7: e6857. Дои:10.7717 / peerj.6857. ЧВК 6500375. PMID 31106066.
- ^ Mykrobe predictor - прогнозирование устойчивости к антибиотикам для S. aureus и M. tuberculosis на основе данных последовательности всего генома
- ^ Быстрые прогнозы устойчивости к антибиотикам на основе данных последовательности генома для Золотистый стафилококк и Микобактерии туберкулеза
- ^ Майкл Мосли против супербактерий
- ^ Mykrobe Predictor github
- ^ Кертис С., Херевард Дж. (29 августа 2017 г.). «От места преступления до зала суда: путешествие образца ДНК». Разговор.
- ^ Морера С., Ларивьер Л., Курцек Дж., Ашке-Зонненборн Ю., Фримонт П.С., Джанин Дж., Рюгер В. (август 2001 г.). «Кристаллические структуры с высоким разрешением бета-глюкозилтрансферазы фага Т4: индуцированное соответствие и эффект связывания субстрата и металла». Журнал молекулярной биологии. 311 (3): 569–77. Дои:10.1006 / jmbi.2001.4905. PMID 11493010.
- ^ Эрлих М., Гама-Соса М.А., Хуанг Л.Х., Миджетт Р.М., Куо К.С., МакКьюн Р.А., Герке С. (апрель 1982 г.). «Количество и распределение 5-метилцитозина в ДНК человека из разных типов тканей клеток». Исследования нуклеиновых кислот. 10 (8): 2709–21. Дои:10.1093 / nar / 10.8.2709. ЧВК 320645. PMID 7079182.
- ^ Эрлих М., Ван Р. Я. (июнь 1981 г.). «5-Метилцитозин в эукариотической ДНК». Наука. 212 (4501): 1350–7. Bibcode:1981Научный ... 212.1350E. Дои:10.1126 / science.6262918. PMID 6262918.
- ^ Song CX, Clark TA, Lu XY, Kislyuk A, Dai Q, Turner SW и др. (Ноябрь 2011 г.). «Чувствительное и специфическое секвенирование одной молекулы 5-гидроксиметилцитозина». Методы природы. 9 (1): 75–7. Дои:10.1038 / nmeth.1779. ЧВК 3646335. PMID 22101853.
- ^ Уотсон Дж. Д., Крик Ф. Х. (1953). «Строение ДНК». Холодная весна Харб. Symp. Quant. Биол. 18: 123–31. Дои:10.1101 / SQB.1953.018.01.020. PMID 13168976.
- ^ Маркс, Л., Путь к секвенированию ДНК: жизнь и работа Фредерика Сенгера.
- ^ Мин Джоу В., Хэгеман Г., Исебаерт М., Фирс В. (май 1972 г.). «Нуклеотидная последовательность гена, кодирующего белок оболочки бактериофага MS2». Природа. 237 (5350): 82–8. Bibcode:1972 год. 237 ... 82J. Дои:10.1038 / 237082a0. PMID 4555447. S2CID 4153893.
- ^ Фирс В., Контрерас Р., Дуеринк Ф., Хегеман Г., Изерентант Д., Меррегерт Дж., Мин Джоу В., Молеманс Ф., Рэймакерс А., Ван ден Берге А., Фолькаерт Г., Исебаерт М. (апрель 1976 г.). «Полная нуклеотидная последовательность РНК бактериофага MS2: первичная и вторичная структура гена репликазы». Природа. 260 (5551): 500–7. Bibcode:1976Натура.260..500F. Дои:10.1038 / 260500a0. PMID 1264203. S2CID 4289674.
- ^ Озсолак Ф., Милош П.М. (февраль 2011 г.). «Секвенирование РНК: достижения, проблемы и возможности». Природа Обзоры Генетика. 12 (2): 87–98. Дои:10.1038 / nrg2934. ЧВК 3031867. PMID 21191423.
- ^ "Профиль факультета Рэя Ву". Корнелл Университет. Архивировано из оригинал 4 марта 2009 г.
- ^ Падманабхан Р., Джей Э., Ву Р. (июнь 1974 г.). «Химический синтез праймера и его использование в анализе последовательности гена лизоцима бактериофага Т4». Труды Национальной академии наук Соединенных Штатов Америки. 71 (6): 2510–4. Bibcode:1974ПНАС ... 71.2510П. Дои:10.1073 / пнас.71.6.2510. ЧВК 388489. PMID 4526223.
- ^ Онага Л.А. (июнь 2014 г.). «Рэй Ву как пятый бизнес: демонстрация коллективной памяти в истории секвенирования ДНК». Исследования по истории и философии науки. Часть C. 46: 1–14. Дои:10.1016 / j.shpsc.2013.12.006. PMID 24565976.
- ^ Ву Р. (1972). «Анализ нуклеотидной последовательности ДНК». Природа Новая Биология. 236 (68): 198–200. Дои:10.1038 / newbio236198a0. PMID 4553110.
- ^ Падманабхан Р., Ву Р. (1972). «Анализ нуклеотидной последовательности ДНК. IX. Использование олигонуклеотидов определенной последовательности в качестве праймеров в анализе последовательности ДНК». Biochem. Биофиз. Res. Сообщество. 48 (5): 1295–302. Дои:10.1016 / 0006-291X (72) 90852-2. PMID 4560009.
- ^ Wu R, Tu CD, Padmanabhan R (1973). «Анализ нуклеотидной последовательности ДНК. XII. Химический синтез и анализ последовательности додекадеоксинуклеотида, который связывается с геном эндолизина бактериофага лямбда». Biochem. Биофиз. Res. Сообщество. 55 (4): 1092–99. Дои:10.1016 / S0006-291X (73) 80007-5. PMID 4358929.
- ^ Джей Э., Бамбара Р., Падманабхан Р., Ву Р. (март 1974 г.). «Анализ последовательности ДНК: общий, простой и быстрый метод секвенирования больших олигодезоксирибонуклеотидных фрагментов путем картирования». Исследования нуклеиновых кислот. 1 (3): 331–53. Дои:10.1093 / nar / 1.3.331. ЧВК 344020. PMID 10793670.
- ^ а б Сэнгер Ф., Никлен С, Колсон А.Р. (декабрь 1977 г.). «Секвенирование ДНК с помощью ингибиторов обрыва цепи». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 74 (12): 5463–77. Bibcode:1977ПНАС ... 74.5463С. Дои:10.1073 / pnas.74.12.5463. ЧВК 431765. PMID 271968.
- ^ а б c Максам AM, Гилберт В. (февраль 1977 г.). «Новый метод секвенирования ДНК». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 74 (2): 560–64. Bibcode:1977ПНАС ... 74..560М. Дои:10.1073 / пнас.74.2.560. ЧВК 392330. PMID 265521.
- ^ Гилберт, В. Секвенирование ДНК и структура гена. Нобелевская лекция, 8 декабря 1980 г.
- ^ Гилберт В., Максам А. (декабрь 1973 г.). «Нуклеотидная последовательность оператора lac». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 70 (12): 3581–84. Bibcode:1973PNAS ... 70.3581G. Дои:10.1073 / pnas.70.12.3581. ЧВК 427284. PMID 4587255.
- ^ Сэнгер Ф., Air GM, Баррелл Б.Г., Браун Н.Л., Колсон А.Р., Фиддес, Калифорния, Хатчисон, Калифорния, Слокомб П.М., Смит М. (февраль 1977 г.). «Нуклеотидная последовательность ДНК бактериофага phi X174». Природа. 265 (5596): 687–95. Bibcode:1977Натура.265..687С. Дои:10.1038 / 265687a0. PMID 870828. S2CID 4206886.
- ^ «Следующий рубеж: вирусы человека» , whatisbiotechnology.org, дата обращения 3 мая 2017.
- ^ Бек С., Поль FM (1984). «Секвенирование ДНК с помощью прямого блоттингового электрофореза». EMBO J. 3 (12): 2905–09. Дои:10.1002 / j.1460-2075.1984.tb02230.x. ЧВК 557787. PMID 6396083.
- ^ Патент США 4631122 (1986)
- ^ Feldmann H, et al. (1994). «Полная последовательность ДНК хромосомы II дрожжей». EMBO J. 13 (24): 5795–809. Дои:10.1002 / j.1460-2075.1994.tb06923.x. ЧВК 395553. PMID 7813418.
- ^ Смит Л. М., Сандерс Дж. З., Кайзер Р. Дж., Хьюз П., Додд С., Коннелл С. Р., Хайнер С., Кент С. Б., Худ Л. Е. (12 июня 1986 г.). «Обнаружение флуоресценции в автоматическом анализе последовательности ДНК». Природа. 321 (6071): 674–79. Bibcode:1986Натура.321..674С. Дои:10.1038 / 321674a0. PMID 3713851. S2CID 27800972.
- ^ Пробер Дж. М., Трейнор Г. Л., Дам Р. Дж., Хоббс Ф. У., Робертсон К. В., Загурски Р. Дж., Кокуцца А. Дж., Дженсен М. А., Баумейстер К. (16 октября 1987 г.). «Система для быстрого секвенирования ДНК с флуоресцентными дидезоксинуклеотидами, завершающими цепь». Наука. 238 (4825): 336–41. Bibcode:1987Наука ... 238..336П. Дои:10.1126 / science.2443975. PMID 2443975.
- ^ Adams MD, Kelley JM, Gocayne JD, Dubnick M, Polymeropoulos MH, Xiao H, Merril CR, Wu A, Olde B, Moreno RF (июнь 1991). «Комплементарное секвенирование ДНК: метки экспрессированной последовательности и проект генома человека». Наука. 252 (5013): 1651–56. Bibcode:1991Научный ... 252.1651A. Дои:10.1126 / science.2047873. PMID 2047873. S2CID 13436211.
- ^ Флейшманн Р.Д., Адамс, доктор медицины, Уайт О., Клейтон Р.А., Киркнесс Э.Ф., Керлаваж А.Р., Булт С.Дж., Могила Д.Ф., Догерти Б.А., Меррик Дж.М. (июль 1995 г.). "Полногеномное случайное секвенирование и сборка Haemophilus influenzae Rd". Наука. 269 (5223): 496–512. Bibcode:1995Научный ... 269..496F. Дои:10.1126 / science.7542800. PMID 7542800.
- ^ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC и др. (Февраль 2001 г.). "Начальная последовательность и анализ человеческого генома" (PDF). Природа. 409 (6822): 860–921. Bibcode:2001Натура.409..860л. Дои:10.1038/35057062. PMID 11237011.
- ^ Вентер Дж. К., Адамс, доктор медицины и др. (Февраль 2001 г.). «Последовательность генома человека». Наука. 291 (5507): 1304–51. Bibcode:2001Научный ... 291.1304V. Дои:10.1126 / science.1058040. PMID 11181995.
- ^ «Эспаснет - Библиографические данные». world.espacenet.com.
- ^ Ронаги М., Карамохамед С., Петтерссон Б., Улен М., Нирен П. (1996). «Секвенирование ДНК в реальном времени с использованием обнаружения высвобождения пирофосфата». Аналитическая биохимия. 242 (1): 84–89. Дои:10.1006 / abio.1996.0432. PMID 8923969.
- ^ а б Кавасима, Эрик Х .; Лоран Фаринелли; Паскаль Майер (12 мая 2005 г.). «Патент: Метод амплификации нуклеиновых кислот». Архивировано из оригинал 22 февраля 2013 г.. Получено 22 декабря 2012.
- ^ Юинг Б., Грин П. (март 1998 г.). «Базовый вызов трассировок автоматического секвенсора с использованием phred. II. Вероятности ошибок». Genome Res. 8 (3): 186–94. Дои:10.1101 / гр. 8.3.186. PMID 9521922.
- ^ «Показатели качества для секвенирования следующего поколения» (PDF). Иллюмина. 31 октября 2011 г.. Получено 8 мая 2018.
- ^ а б Бреннер С., Джонсон М., Бриджем Дж., Голда Дж., Ллойд Д.Х., Джонсон Д., Луо С., МакКарди С., Фой М., Юэн М., Рот Р., Джордж Д., Элетр С., Альбрехт Дж., Вермаас Е., Уильямс С. Р., Мун К. , Бурчам Т., Паллас М., ДюБридж Р. Б., Киршнер Дж., Фирон К., Мао Дж., Коркоран К. (2000). «Анализ экспрессии генов путем массового параллельного секвенирования сигнатур (MPSS) на массивах микрогранул». Природа Биотехнологии. 18 (6): 630–34. Дои:10.1038/76469. PMID 10835600. S2CID 13884154.
- ^ Сэнгер Ф., Колсон А.Р. (май 1975 г.). «Экспресс-метод определения последовательностей в ДНК путем примированного синтеза с ДНК-полимеразой». J. Mol. Биол. 94 (3): 441–48. Дои:10.1016/0022-2836(75)90213-2. PMID 1100841.
- ^ Веттерстранд, Крис. «Стоимость секвенирования ДНК: данные программы секвенирования генома NHGRI (GSP)». Национальный институт исследования генома человека. Получено 30 мая 2013.
- ^ Перепел М.А., Гу Й, Свердлов Х, Мэйхо М. (2012). «Оценка и оптимизация препаративных полуавтоматических систем электрофореза для подготовки библиотеки Illumina». Электрофорез. 33 (23): 3521–28. Дои:10.1002 / elps.201200128. PMID 23147856. S2CID 39818212.
- ^ Duhaime MB, Deng L, Poulos BT, Sullivan MB (2012). «К количественной метагеномике диких вирусов и других образцов ДНК со сверхнизкими концентрациями: строгая оценка и оптимизация метода амплификации линкера». Environ. Микробиол. 14 (9): 2526–37. Дои:10.1111 / j.1462-2920.2012.02791.x. ЧВК 3466414. PMID 22713159.
- ^ Петерсон Б.К., Вебер Дж. Н., Кей Э. Х., Фишер Х. С., Хоэкстра Х. Э. (2012). «Двойной дайджест RADseq: недорогой метод обнаружения de novo SNP и генотипирования у модельных и немодельных видов». PLOS ONE. 7 (5): e37135. Bibcode:2012PLoSO ... 737135P. Дои:10.1371 / journal.pone.0037135. ЧВК 3365034. PMID 22675423.
- ^ Уильямс Р., Пейсайович С.Г., Миллер О.Дж., Магдасси С., Тауфик Д.С., Гриффитс А.Д. (2006). «Амплификация сложных библиотек генов методом эмульсионной ПЦР». Методы природы. 3 (7): 545–50. Дои:10.1038 / nmeth896. PMID 16791213. S2CID 27459628.
- ^ а б Маргулис М., Эгхолм М. и др. (Сентябрь 2005 г.). «Секвенирование генома в открытых микропроцессорных пиколитерных реакторах высокой плотности». Природа. 437 (7057): 376–80. Bibcode:2005Натура 437..376М. Дои:10.1038 / природа03959. ЧВК 1464427. PMID 16056220.
- ^ Шендуре Дж., Поррека Дж. Дж., Реппас Н. Б., Лин Х, МакКатчеон Дж. П., Розенбаум А. М., Ван М. Д., Чжан К., Митра Р. Д., Чёрч Г. М. (2005). «Точное мультиплексное секвенирование полонии эволюционировавшего бактериального генома». Наука. 309 (5741): 1728–32. Bibcode:2005Наука ... 309.1728S. Дои:10.1126 / science.1117389. PMID 16081699. S2CID 11405973.
- ^ «Прикладные биосистемы - файл не найден (ошибка 404)». 16 мая 2008 г. Архивировано с оригинал 16 мая 2008 г.
- ^ Гудвин С., Макферсон Дж. Д., Маккомби В. Р. (май 2016 г.). «Достигнув совершеннолетия: десять лет технологий секвенирования следующего поколения». Природа Обзоры Генетика. 17 (6): 333–51. Дои:10.1038 / nrg.2016.49. PMID 27184599. S2CID 8295541.
- ^ Staden R (11 июня 1979 г.). «Стратегия секвенирования ДНК с использованием компьютерных программ». Исследования нуклеиновых кислот. 6 (7): 2601–10. Дои:10.1093 / nar / 6.7.2601. ЧВК 327874. PMID 461197.
- ^ де Magalhães JP, Finch CE, Janssens G (2010). «Секвенирование нового поколения в исследованиях старения: новые приложения, проблемы, подводные камни и возможные решения». Обзоры исследований старения. 9 (3): 315–23. Дои:10.1016 / j.arr.2009.10.006. ЧВК 2878865. PMID 19900591.
- ^ Града А (август 2013 г.). «Секвенирование следующего поколения: методология и применение». J Invest Dermatol. 133 (8): e11. Дои:10.1038 / jid.2013.248. PMID 23856935.
- ^ Зал N (май 2007 г.). «Передовые технологии секвенирования и их более широкое влияние на микробиологию». J. Exp. Биол. 210 (Pt 9): 1518–25. Дои:10.1242 / jeb.001370. PMID 17449817.

- ^ Церковь GM (Январь 2006 г.). «Геномы для всех». Sci. Являюсь. 294 (1): 46–54. Bibcode:2006SciAm.294a..46C. Дои:10.1038 / scientificamerican0106-46. PMID 16468433.(требуется подписка)
- ^ а б c Schuster SC (январь 2008 г.). «Секвенирование следующего поколения меняет сегодняшнюю биологию». Nat. Методы. 5 (1): 16–18. Дои:10.1038 / nmeth1156. PMID 18165802. S2CID 1465786.
- ^ Кальб, Гилберт; Моксли, Роберт (1992). Массовые параллельные, оптические и нейронные вычисления в США. IOS Press. ISBN 978-90-5199-097-3.[страница нужна]
- ^ десять Bosch JR, Grody WW (2008). «Идти в ногу со следующим поколением». Журнал молекулярной диагностики. 10 (6): 484–92. Дои:10.2353 / jmoldx.2008.080027. ЧВК 2570630. PMID 18832462.

- ^ Такер Т., Марра М., Фридман Дж. М. (2009). «Массивно параллельное секвенирование: следующий большой шаг в генетической медицине». Американский журнал генетики человека. 85 (2): 142–54. Дои:10.1016 / j.ajhg.2009.06.022. ЧВК 2725244. PMID 19679224.

- ^ а б Стрэйтон Дж., Фри Т, Сойер А., Мартин Дж. (Февраль 2019 г.). «От секвенирования по Сэнгеру до баз данных генома и не только». Биотехнологии. Будущее науки. 66 (2): 60–63. Дои:10.2144 / btn-2019-0011. PMID 30744413.
- ^ Перепел М.А., Смит М., Коупленд П., Отто Т.Д., Харрис С.Р., Коннор Т.Р., Бертони А., Свердлоу Х.П., Гу Y (1 января 2012 г.). «Рассказ о трех платформах секвенирования следующего поколения: сравнение секвенсоров Ion Torrent, Pacific Biosciences и illumina MiSeq». BMC Genomics. 13 (1): 341. Дои:10.1186/1471-2164-13-341. ЧВК 3431227. PMID 22827831.

- ^ Лю Л., Ли И, Ли С., Ху Н, Хе И, Понг Р., Лин Д., Лу Л., Ло М. (1 января 2012 г.). «Сравнение систем секвенирования нового поколения». Журнал биомедицины и биотехнологии. 2012: 251364. Дои:10.1155/2012/251364. ЧВК 3398667. PMID 22829749.

- ^ а б c «Новое программное обеспечение, полимераза для увеличения пропускной способности и доступности системы Sequel - PacBio». 7 марта 2018.
- ^ «После года испытаний два первых клиента PacBio ожидают более регулярного использования RS Sequencer в 2012 году». GenomeWeb. 10 января 2012 г.(требуется регистрация)
- ^ Inc., Pacific Biosciences (2013). «Pacific Biosciences представляет новую химию с увеличенной длиной считывания для обнаружения новых особенностей в последовательности ДНК и продвинутых исследований генома крупных организмов».
- ^ Чин С.С., Александр Д.Х., Маркс П., Кламмер А.А., Дрейк Дж., Хайнер С., Клам А., Коупленд А., Хаддлстон Дж., Эйхлер Е.Э., Тернер С.В., Корлах Дж. (2013). «Негибридные, готовые сборки микробного генома на основе данных секвенирования SMRT». Nat. Методы. 10 (6): 563–69. Дои:10.1038 / nmeth.2474. PMID 23644548. S2CID 205421576.
- ^ а б "Сборка бактериального генома de novo: проблема решенная?". 5 июля 2013 г.
- ^ Раско Д.А., Вебстер Д.Р., Сахл Дж. В., Башир А., Бойзен Н., Шойц Ф., Паксинос Э., Себра Р., Чин С. С., Илиопулос Д., Кламмер А., Пелусо П., Ли Л., Кислюк А. О., Буллард Дж., Касарскис А., Ван С. , Eid J, Rank D, Redman JC, Steyert SR, Frimodt-Møller J, Struve C, Petersen AM, Krogfelt KA, Nataro JP, Schadt EE, Waldor MK (25 августа 2011 г.). «Истоки штамма, вызвавшего вспышку гемолитико-уремического синдрома в Германии». N Engl J Med. 365 (8): 709–17. Дои:10.1056 / NEJMoa1106920. ЧВК 3168948. PMID 21793740.

- ^ Тран Б., Браун А.М., Бедард П.Л., Винквист Э., Госс Г.Д., Хотте С.Дж., Уэлч С.А., Хирте Х.В., Чжан Т., Штейн Л.Д., Ферретти В, Ватт С, Цзяо В, Нг К, Гхай С., Шоу П., Петрочелли Т., Хадсон TJ, Neel BG, Onetto N, Siu LL, McPherson JD, Kamel-Reid S, Dancey JE (1 января 2012 г.). «Возможность секвенирования раковых генов следующего поколения в реальном времени, связанных с ответом на лекарства: результаты клинических испытаний». Int. J. Рак. 132 (7): 1547–55. Дои:10.1002 / ijc.27817. PMID 22948899. S2CID 72705.(требуется подписка)
- ^ Мюррей И.А., Кларк Т.А., Морган Р.Д., Бойтано М., Антон Б.П., Луонг К., Фоменков А., Тернер С.В., Корлах Дж., Робертс Р.Дж. (2 октября 2012 г.). «Метиломы шести бактерий». Исследования нуклеиновых кислот. 40 (22): 11450–62. Дои:10.1093 / нар / gks891. ЧВК 3526280. PMID 23034806.
- ^ «Комплект Ion 520 и Ion 530 ExT - Шеф-повар - Thermo Fisher Scientific». www.thermofisher.com.
- ^ «Архивная копия». Архивировано из оригинал 30 марта 2018 г.. Получено 29 марта 2018.CS1 maint: заархивированная копия как заголовок (связь)
- ^ ван Влит А.Х. (1 января 2010 г.). «Секвенирование микробных транскриптомов нового поколения: проблемы и возможности». Письма о микробиологии FEMS. 302 (1): 1–7. Дои:10.1111 / j.1574-6968.2009.01767.x. PMID 19735299.

- ^ «БГИ и МГИСЭК». en.mgitech.cn. Получено 5 июля 2018.
- ^ а б Хуан Ю.Ф., Чен С.К., Чианг Ю.С., Чен Т.Х., Чиу КП (2012). «Палиндромная последовательность препятствует механизму секвенирования путем лигирования». BMC Системная биология. 6 Приложение 2: S10. Дои:10.1186 / 1752-0509-6-S2-S10. ЧВК 3521181. PMID 23281822.
- ^ Свободный, Мэтью; Ракьян, Вардхман; Холмс, Надин; Пейн, Александр (3 мая 2018 г.). «Наблюдение за китами с BulkVis: графический просмотрщик для файлов Oxford Nanopore bulk fast5». bioRxiv 10.1101/312256.
- ^ «Продажи PacBio начинают расти по мере того, как компания расширяет возможности продукта».
- ^ «Мир Био-ИТ». www.bio-itworld.com.
- ^ «PacBio запускает высокопроизводительную и недорогую систему секвенирования одиночных молекул».
- ^ Кларк Дж., Ву ХК, Джаясингхе Л., Патель А., Рид С., Бейли Х. (апрель 2009 г.). «Непрерывная идентификация оснований для секвенирования одномолекулярных нанопор ДНК». Природа Нанотехнологии. 4 (4): 265–70. Bibcode:2009НатНа ... 4..265С. Дои:10.1038 / nnano.2009.12. PMID 19350039.
- ^ а б Дела Торре Р., Ларкин Дж., Певица А., Меллер А. (2012). «Изготовление и характеристика массивов твердотельных нанопор для высокопроизводительного секвенирования ДНК». Нанотехнологии. 23 (38): 385308. Bibcode:2012Nanot..23L5308D. Дои:10.1088/0957-4484/23/38/385308. ЧВК 3557807. PMID 22948520.
- ^ а б Патхак Б., Лофас Х., Прасонгкит Дж., Григорьев А., Ахуджа Р., Шайхер Р.Х. (2012). «Золотые электроды с двойными функционализированными нанопорами для быстрого секвенирования ДНК». Письма по прикладной физике. 100 (2): 023701. Bibcode:2012АпФЛ.100б3701П. Дои:10.1063/1.3673335.
- ^ Корлач Дж, Маркс П.Дж., Цицерон Р.Л., Грей Дж.Дж., Мерфи Д.Л., Ройтман Д.Б., Фам Т.Т., Отто Г.А., Фокет М., Тернер С.В. (2008). «Селективная пассивация алюминием для направленной иммобилизации одиночных молекул ДНК-полимеразы в волноводных наноструктурах с нулевой модой». Труды Национальной академии наук. 105 (4): 1176–81. Bibcode:2008ПНАС..105.1176К. Дои:10.1073 / pnas.0710982105. ЧВК 2234111. PMID 18216253.
- ^ а б Шендуре Дж., Поррека Дж. Дж., Реппас Н. Б., Лин Х, МакКатчеон Дж. П., Розенбаум А. М., Ван М. Д., Чжан К., Митра Р. Д., Церковный генеральный директор (9 сентября 2005 г.). «Точное мультиплексное секвенирование полонии эволюционировавшего бактериального генома». Наука. 309 (5741): 1728–32. Bibcode:2005Sci ... 309.1728S. Дои:10.1126 / science.1117389. PMID 16081699. S2CID 11405973.
- ^ Бентли Д. Р., Баласубраманиан С. и др. (2008). «Точное секвенирование всего генома человека с использованием химии обратимых терминаторов». Природа. 456 (7218): 53–59. Bibcode:2008Натура 456 ... 53Б. Дои:10.1038 / природа07517. ЧВК 2581791. PMID 18987734.
- ^ Канард Б., Сарфати С. (13 октября 1994 г.), Новые производные, используемые для секвенирования нуклеиновых кислот, получено 9 марта 2016
- ^ Канард Б., Сарфати Р.С. (октябрь 1994 г.). «Флуоресцентные субстраты ДНК-полимеразы с обратимыми 3'-метками». Ген. 148 (1): 1–6. Дои:10.1016/0378-1119(94)90226-7. PMID 7523248.
- ^ Мардис ER (2008). «Методы секвенирования ДНК нового поколения». Анну Рев Геном Хум Генет. 9: 387–402. Дои:10.1146 / annurev.genom.9.081307.164359. PMID 18576944.
- ^ а б c Drmanac R, Sparks AB, Callow MJ, Halpern AL, Burns NL, Kermani BG и др. (Январь 2010 г.). «Секвенирование генома человека с использованием несвязанного основания считывает самособирающиеся наномассивы ДНК». Наука. 327 (5961): 78–81. Bibcode:2010Sci ... 327 ... 78D. Дои:10.1126 / science.1181498. PMID 19892942. S2CID 17309571.
- ^ brandonvd. «О нас - Полная геномика». Полная геномика. Получено 2 июля 2018.
- ^ а б Хуанг Дж, Лян Х, Сюань И, Гэн Ц, Ли И, Лу Х и др. (Май 2017). «Эталонный набор данных генома человека секвенатора BGISEQ-500». GigaScience. 6 (5): 1–9. Дои:10.1093 / gigascience / gix024. ЧВК 5467036. PMID 28379488.
- ^ Валуев А., Итикава Дж., Тонтхат Т., Стюарт Дж., Ранад С., Пекхэм Х., Зенг К., Малек Дж. А., Коста Дж., МакКернан К., Сидоу А., Файер А., Джонсон С. М. (июль 2008 г.). «Карта положения нуклеосом C. elegans с высоким разрешением демонстрирует отсутствие универсального позиционирования, определяемого последовательностью». Genome Res. 18 (7): 1051–63. Дои:10.1101 / гр.076463.108. ЧВК 2493394. PMID 18477713.
- ^ Раск Н (2011). «Торренты последовательности». Нат методы. 8 (1): 44. Дои:10.1038 / nmeth.f.330. S2CID 41040192.
- ^ а б Drmanac R, Sparks AB и др. (2010). «Секвенирование генома человека с использованием считываний несвязанных оснований в самоорганизующихся наноразмерных массивах ДНК». Наука. 327 (5961): 78–81. Bibcode:2010Sci ... 327 ... 78D. Дои:10.1126 / science.1181498. PMID 19892942. S2CID 17309571.
- ^ Porreca GJ (2010). «Секвенирование генома на наношарах». Природа Биотехнологии. 28 (1): 43–44. Дои:10.1038 / nbt0110-43. PMID 20062041. S2CID 54557996.
- ^ Полная геномика Пресс-релиз, 2010 г.
- ^ "Система секвенирования генов / генетического анализатора HeliScope: Helicos BioSciences". 2 ноября 2009 г. Архивировано с оригинал 2 ноября 2009 г.
- ^ Томпсон Дж. Ф., Штейнманн К. Э. (октябрь 2010 г.). Секвенирование отдельной молекулы с помощью системы генетического анализа HeliScope. Текущие протоколы в молекулярной биологии. Глава 7. С. Unit7.10. Дои:10.1002 / 0471142727.mb0710s92. ISBN 978-0471142720. ЧВК 2954431. PMID 20890904.
- ^ «Техническое объяснение tSMS SeqLL». SeqLL. Архивировано из оригинал 8 августа 2014 г.. Получено 9 августа 2015.
- ^ Хизер, Джеймс М .; Цепь, Бенджамин (январь 2016 г.). «Последовательность секвенаторов: история секвенирования ДНК». Геномика. 107 (1): 1–8. Дои:10.1016 / j.ygeno.2015.11.003. ISSN 1089-8646. ЧВК 4727787. PMID 26554401.
- ^ Сара Эль-Метвалли; Усама М. Уда; Мохамед Хельми (2014). «Новые горизонты в секвенировании следующего поколения». Технологии секвенирования нового поколения и проблемы сборки последовательностей. Технологии секвенирования нового поколения и проблемы сборки последовательностей, Springer Briefs in Systems Biology Volume 7. SpringerBriefs по системной биологии. 7. С. 51–59. Дои:10.1007/978-1-4939-0715-1_6. ISBN 978-1-4939-0714-4.
- ^ а б Ярмарка РБ, Хлыстов А., Портной Т.Д., Иванов В., Эванс Р.Д., Сринивасан В., Памула В.К., Поллак М.Г., Гриффин ПБ, Чжоу Дж. (Январь 2007 г.). «Химические и биологические применения цифровых микрофлюидных устройств». Дизайн и тестирование компьютеров IEEE. 24 (1): 10–24. CiteSeerX 10.1.1.559.1440. Дои:10.1109 / MDT.2007.8. HDL:10161/6987. S2CID 10122940.
- ^ а б Болес Д., Бентон Дж. Л., Сью Дж. Дж., Леви М. Х., Твар П. К., Сандал М. А. и др. (Ноябрь 2011 г.). «Пиросеквенирование на основе капель с использованием цифровой микрофлюидики». Аналитическая химия. 83 (22): 8439–47. Дои:10.1021 / ac201416j. ЧВК 3690483. PMID 21932784.
- ^ Зилионис Р., Наинис Дж., Верес А., Савова В., Земмур Д., Кляйн А.М., Мазутис Л. (январь 2017 г.). «Штрих-кодирование одной клетки и секвенирование с использованием капельной микрофлюидики». Протоколы природы. 12 (1): 44–73. Дои:10.1038 / nprot.2016.154. PMID 27929523. S2CID 767782.
- ^ "Гарвардская группа нанопор". Mcb.harvard.edu. Архивировано из оригинал 21 февраля 2002 г.. Получено 15 ноября 2009.
- ^ «Секвенирование нанопор может снизить затраты на анализ ДНК».
- ^ Патент США 20060029957, ZS Genetics, "Системы и методы анализа полимеров нуклеиновых кислот и родственных компонентов", выпущен 14 июля 2005 г.
- ^ Сюй М., Фудзита Д., Ханагата Н. (декабрь 2009 г.). «Перспективы и проблемы новых технологий секвенирования одномолекулярной ДНК». Маленький. 5 (23): 2638–49. Дои:10.1002 / smll.200900976. PMID 19904762.
- ^ Шадт Э., Тернер С, Касарскис А (2010). «Окно в секвенирование третьего поколения». Молекулярная генетика человека. 19 (R2): R227–40. Дои:10,1093 / hmg / ddq416. PMID 20858600.
- ^ Сюй М., Эндрес Р.Г., Аракава Ю. (2007). «Электронные свойства основ ДНК». Маленький. 3 (9): 1539–43. Дои:10.1002 / smll.200600732. PMID 17786897.
- ^ Ди Вентра М (2013). «Быстрое секвенирование ДНК электрическими средствами на несколько дюймов ближе». Нанотехнологии. 24 (34): 342501. Bibcode:2013Nanot..24H2501D. Дои:10.1088/0957-4484/24/34/342501. PMID 23899780.
- ^ Оширо Т., Мацубара К., Цуцуи М., Фурухаши М., Танигучи М., Каваи Т. (2012). «Электрическое случайное переупорядочение одной молекулы ДНК и РНК». Научный представитель. 2: 501. Bibcode:2012НатСР ... 2E.501O. Дои:10.1038 / srep00501. ЧВК 3392642. PMID 22787559.
- ^ Ханна Г.Дж., Джонсон В.А., Курицкес Д.Р., Ричман Д.Д., Мартинес-Пикадо Дж., Саттон Л., Хейзелвуд Д.Д., Д'Акуила, RT (1 июля 2000 г.). «Сравнение секвенирования путем гибридизации и циклического секвенирования для генотипирования обратной транскриптазы вируса иммунодефицита человека типа 1». J. Clin. Микробиол. 38 (7): 2715–21. Дои:10.1128 / JCM.38.7.2715-2721.2000. ЧВК 87006. PMID 10878069.
- ^ а б Морей М., Фернандес-Мармиесс А., Кастиньейрас Д., Фрага Дж. М., Кус М. Л., Кочо Дж. А. (2013). «Взгляд в прошлое, настоящее и будущее секвенирования ДНК». Молекулярная генетика и метаболизм. 110 (1–2): 3–24. Дои:10.1016 / j.ymgme.2013.04.024. PMID 23742747.
- ^ Цинь Y, Шнайдер TM, Бреннер MP (2012). Гибас С (ред.). «Секвенирование путем гибридизации длинных мишеней». PLOS ONE. 7 (5): e35819. Bibcode:2012PLoSO ... 735819Q. Дои:10.1371 / journal.pone.0035819. ЧВК 3344849. PMID 22574124.
- ^ Эдвардс-младший, Рупарел Х, Джу Дж (2005). «Масс-спектрометрическое секвенирование ДНК». Мутационные исследования. 573 (1–2): 3–12. Дои:10.1016 / j.mrfmmm.2004.07.021. PMID 15829234.
- ^ Холл Т.А., Бадоуле Б., Цзян И., Блин Л., Эшу М., Саннес-Лоури К.А., Сампат Р., Дрейдер Дж. Дж., Ханнис Дж. К., Харрелл П., Самант В., Уайт Н., Эккер Д. Д., Хофстадлер С. А. (2005) «Анализ основного состава митохондриальной ДНК человека с использованием масс-спектрометрии с ионизацией электрораспылением: новый инструмент для идентификации и дифференциации людей». Аналитическая биохимия. 344 (1): 53–69. Дои:10.1016 / j.ab.2005.05.028. PMID 16054106.
- ^ Ховард Р., Энчева В., Томсон Дж., Бач К., Чан Ю. Т., Коуэн С., Дебенхэм П., Диксон А., Краузе Дж. Ю, Кришан Д., Мур Д., Мур В., Оджо М., Родригес С., Стокс П., Уокер Дж., Циммерманн В. , Бараллон Р. (15 июня 2011 г.). «Сравнительный анализ митохондриальной ДНК человека из образцов костей времен Первой мировой войны с помощью секвенирования ДНК и масс-спектрометрии ESI-TOF». Forensic Science International: генетика. 7 (1): 1–9. Дои:10.1016 / j.fsigen.2011.05.009. PMID 21683667.
- ^ Monforte JA, Becker CH (1 марта 1997 г.). «Высокопроизводительный анализ ДНК методом времяпролетной масс-спектрометрии». Природа Медицина. 3 (3): 360–62. Дои:10,1038 / нм0397-360. PMID 9055869. S2CID 28386145.
- ^ Берес С.Б., Кэрролл Р.К., Ши П.Р., Ситкевич И., Мартинес-Гутьеррес Дж. К., Лоу ДЭ, МакГир А., Вилли Б.М., Грин К, Тиррелл Г.Дж., Голдман Т.Д., Фельдгарден М., Биррен Б.В., Фофанов Ю., Боос Дж., Уитон В. Honisch C, Musser JM (8 февраля 2010 г.). «Молекулярная сложность последовательных бактериальных эпидемий после деконволюции сравнительной патогеномики». Труды Национальной академии наук. 107 (9): 4371–76. Bibcode:2010PNAS..107.4371B. Дои:10.1073 / pnas.0911295107. ЧВК 2840111. PMID 20142485.
- ^ Кан Ч.В., Фредлейк С.П., Доэрти Э.А., Бэррон А.Э. (1 ноября 2004 г.). «Секвенирование ДНК и генотипирование в миниатюрных системах электрофореза». Электрофорез. 25 (21–22): 3564–88. Дои:10.1002 / elps.200406161. PMID 15565709. S2CID 4851728.
- ^ Чен Й.Дж., Роллер Е.Е., Хуан Х (2010). «Секвенирование ДНК денатурацией: экспериментальное подтверждение концепции со встроенным жидкостным устройством». Лаборатория на чипе. 10 (9): 1153–59. Дои:10.1039 / b921417h. ЧВК 2881221. PMID 20390134.
- ^ Белл, округ Колумбия, Томас В.К., Муртаг К.М., Дионн Калифорния, Грэм А.С., Андерсон Дж. Э., Гловер В. Р. (9 октября 2012 г.). «Идентификация оснований ДНК с помощью электронной микроскопии». Микроскопия и микроанализ. 18 (5): 1049–53. Bibcode:2012MiMic..18.1049B. Дои:10.1017 / S1431927612012615. PMID 23046798.
- ^ Pareek CS, Smoczynski R, Tretyn A (ноябрь 2011 г.). «Технологии секвенирования и секвенирование генома». Журнал прикладной генетики. 52 (4): 413–35. Дои:10.1007 / s13353-011-0057-х. ЧВК 3189340. PMID 21698376.
- ^ Pareek CS, Смочинский Р., Третьин А (2011). «Технологии секвенирования и секвенирование генома». Журнал прикладной генетики. 52 (4): 413–35. Дои:10.1007 / s13353-011-0057-х. ЧВК 3189340. PMID 21698376.
- ^ Fujimori S, Hirai N, Ohashi H, Masuoka K, Nishikimi A, Fukui Y, Washio T, Oshikubo T., Yamashita T., Miyamoto-Sato E (2012). «Секвенирование нового поколения в сочетании с технологией бесклеточного отображения для высокопроизводительного получения надежных данных для взаимодействия». Научные отчеты. 2: 691. Bibcode:2012НатСР ... 2Е.691Ф. Дои:10.1038 / srep00691. ЧВК 3466446. PMID 23056904.
- ^ Харберс М (2008). «Текущее состояние клонирования кДНК». Геномика. 91 (3): 232–42. Дои:10.1016 / j.ygeno.2007.11.004. PMID 18222633.
- ^ Альберти А., Белсер С., Энгелен С., Бертран Л., Орвен С., Бринас Л., Круод С. и др. (2014). «Сравнение методов подготовки библиотек показывает их влияние на интерпретацию метатранскриптомических данных». BMC Genomics. 15: 912–12. Дои:10.1186/1471-2164-15-912. ЧВК 4213505. PMID 25331572.
- ^ «Масштабируемая оценка качества нуклеиновых кислот для подготовки библиотеки секвенирования нового поколения Illumina» (PDF). Получено 27 декабря 2017.
- ^ "Archon Genomics XPRIZE". Archon Genomics XPRIZE.
- ^ «Информация о гранте». Национальный институт исследования генома человека (NHGRI).
- ^ Северин Дж., Лизио М., Харшбаргер Дж., Кавадзи Х., Дауб КО, Хаяшизаки Ю., Бертин Н., Форрест А. Р. (2014). «Интерактивная визуализация и анализ крупномасштабных наборов данных секвенирования с использованием ZENBU». Nat. Биотехнология. 32 (3): 217–19. Дои:10.1038 / nbt.2840. PMID 24727769. S2CID 26575621.
- ^ Шмилович А., Бен-Гал И. (2007). «Использование модели VOM для реконструкции потенциальных кодирующих областей в последовательностях EST» (PDF). Вычислительная статистика. 22 (1): 49–69. Дои:10.1007 / s00180-007-0021-8. S2CID 2737235.
- ^ Дель Фаббро С, Скалабрин С, Морганте М, Джордж ФМ (2013). «Обширная оценка влияния обрезки показаний на анализ данных Illumina NGS». PLOS ONE. 8 (12): e85024. Bibcode:2013PLoSO ... 885024D. Дои:10.1371 / journal.pone.0085024. ЧВК 3871669. PMID 24376861.
- ^ Мартин, Марсель (2 мая 2011 г.). «Cutadapt удаляет последовательности адаптеров из операций чтения с высокой пропускной способностью». EMBnet.journal. 17 (1): 10. Дои:10.14806 / ej.17.1.200.
- ^ Смедс Л., Кюнстнер А. (19 октября 2011 г.). «ConDeTri - зависимый от содержимого триммер чтения для данных Illumina». PLOS ONE. 6 (10): e26314. Bibcode:2011PLoSO ... 626314S. Дои:10.1371 / journal.pone.0026314. ЧВК 3198461. PMID 22039460.
- ^ Prezza N, Del Fabbro C, Vezzi F, De Paoli E, Policriti A (2012). ERNE-BS5: Выравнивание обработанных BS последовательностей множественными ударами по 5-буквенному алфавиту. Материалы конференции ACM по биоинформатике, компьютерной биологии и биомедицине. 12. С. 12–19. Дои:10.1145/2382936.2382938. ISBN 9781450316705. S2CID 5673753.
- ^ Шмидер Р., Эдвардс Р. (март 2011 г.). «Контроль качества и предварительная обработка наборов метагеномных данных». Биоинформатика. 27 (6): 863–4. Дои:10.1093 / биоинформатика / btr026. ЧВК 3051327. PMID 21278185.
- ^ Болджер А.М., Лозе М., Усадель Б (август 2014 г.). «Trimmomatic: гибкий триммер для данных последовательности Illumina». Биоинформатика. 30 (15): 2114–20. Дои:10.1093 / биоинформатика / btu170. ЧВК 4103590. PMID 24695404.
- ^ Кокс МП, Петерсон Д.А., Биггс П.Дж. (сентябрь 2010 г.). «SolexaQA: Краткая оценка качества данных секвенирования второго поколения Illumina». BMC Bioinformatics. 11 (1): 485. Дои:10.1186/1471-2105-11-485. ЧВК 2956736. PMID 20875133.
- ^ Мюррей TH (январь 1991 г.). «Этические вопросы исследования генома человека». Журнал FASEB. 5 (1): 55–60. Дои:10.1096 / fasebj.5.1.1825074. PMID 1825074. S2CID 20009748.
- ^ а б c Робертсон Дж. А. (август 2003 г.). «Геном за 1000 долларов: этические и юридические вопросы при секвенировании всего генома людей». Американский журнал биоэтики. 3 (3): W – IF1. Дои:10.1162/152651603322874762. PMID 14735880. S2CID 15357657.
- ^ а б Хендерсон, Марк (9 сентября 2013 г.). «Секвенирование генома человека: настоящие этические дилеммы». Хранитель. Получено 20 мая 2015.
- ^ Хармон, Эми (24 февраля 2008 г.). «Страхи заставляют многих избегать тестов ДНК». Нью-Йорк Таймс. Получено 20 мая 2015.
- ^ Заявление о политике администрации, Исполнительная канцелярия президента, Управление по вопросам управления и бюджета, 27 апреля 2007 г.
- ^ Национальный институт исследования генома человека (21 мая 2008 г.). «Президент Буш подписывает Закон о недискриминации в области генетической информации 2008 года». Получено 17 февраля 2014.
- ^ Бейкер, Моня. «Комиссия по этике США сообщает о секвенировании ДНК и конфиденциальности». Природа Новый блог. Получено 20 мая 2015.
- ^ «Конфиденциальность и прогресс в секвенировании всего генома» (PDF). Президентская комиссия по изучению вопросов биоэтики. Архивировано из оригинал (PDF) 12 июня 2015 г.. Получено 20 мая 2015.
- ^ Гольденберг А.Дж., Шарп Р.Р. (февраль 2012 г.). «Этические опасности и программные проблемы геномного скрининга новорожденных». JAMA. 307 (5): 461–2. Дои:10.1001 / jama.2012.68. ЧВК 3868436. PMID 22298675.
- ^ Хьюз, Вирджиния (7 января 2013 г.). «Пора перестать зацикливаться на опасности генетической информации». Slate Magazine. Получено 22 мая 2015.
- ^ а б Bloss CS, Schork NJ, Topol EJ (февраль 2011 г.). «Эффект прямого геномного профилирования для оценки риска заболевания». Медицинский журнал Новой Англии. 364 (6): 524–34. Дои:10.1056 / NEJMoa1011893. ЧВК 3786730. PMID 21226570.
- ^ Рохман, Бонни (25 октября 2012 г.). "Что ваш доктор не говорит вам о вашей ДНК". Time.com. Получено 22 мая 2015.
внешняя ссылка
| Библиотечные ресурсы о Секвенирование ДНК |
| В Викиучебнике есть книга на следующие темы: Секвенирование следующего поколения (NGS) |